Estructura del ADN y su replicación
-
Upload
jose-alfredo-cervantes -
Category
Documents
-
view
233 -
download
3
description
Transcript of Estructura del ADN y su replicación

José Alfredo Cervantes Sánchez
00096493 / 10409
Estructura del DNA y su replicación
1. DNA y su extructura
El DNA y el RNA son polímeros (múltiples unidades químicas) lineales compuestos por
monómeros (unidades químicas aisladas) denominados nucleótidos. El RNA varía en
longitud desde menos de 100 a varios miles de nucleótidos, en cambio las moléculas de
DNA pueden contener hasta varios cientos de millones de nucleótidos.
Nucleótidos.
Los nucleótidos tienen una estructura en común; un grupo fosfato unido por un enlace
fosfoester a una pentosa (una molécula de azúcar de cinco carbonos), que, a su vez, se
une a una base nitrogenada.
Bases nitrogenadas
Entre DNA y RNA hay ciertas diferencias:
DNA RNA
La pentosa es desoxirribosa La pentosa es ribosa
Solo aquí se presenta timina Solo aquí se presenta uracilo
Cadena doble Cadena sencilla
Los dos presentan adenina, citosina y guanina
Nucleósidos.
Son combinaciones de una base con un azúcar sin fosfato. Los nucleótidos son
nucleósidos con uno, dos o tres grupos fosfato, dependiendo del número se clasifican en
monofosfatos, difosfatos o trifosfatos.
Nombres de bases dependiendo si tienen o no el grupo fosfato:
Par de anillos
fucionados
Un solo anillo

2
BASES NUCLEÓSIDO NUCLEÓTIDO
A Adenina adenosina adenosina fosfato
(ac. adenílico)
C citosina citidina citidina fosfato
(ac. citidílico)
G guanina guanosina guanosina fosfato
(ac. guanidílico)
T timina timidina timidina fosfato
(ac. timidílico)
Ácidos nucleicos.
Son nucleótidos polimerizados, el grupo hidroxilo fijado al carbono 3’ del azúcar de un
nucleótido forma un enlace ester con el fosfato de otro nucleótido, con eliminación de agua,
la unión entre nucleótidos se denomina enlace fosfodiester.
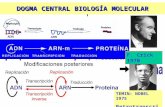
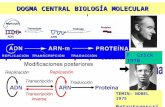
Dogma central de la biología molecular:
El DNA la molécula de la vida
Consta de dos hebras de polinucleótidos asociadas que se entrelazan entre sí en el
espacio, para formar una estructura que se suele describir como una doble hélice.
Las dos columnas vertebrales de azúcar-fosfato se ubican en la parte externa de la doble
hélice y las bases se proyectan hacia el interior.
Las bases adyacentes de cada hebra se apilan una sobre otra en planos paralelos. Las
dos hebras tienen orientación antiparalela; o sea que sus direcciones 5’ => 3’ son opuestas.
Las hebras mantienen un registro exacto debido al apareamiento regular de las bases entre
ambas hebras;
-Adenosina se aparea con Timina por medio de dos enlaces de hidrogeno.

3
-Guanina se aparea con Citosina por medio de tres enlaces de hidrogeno.
*Puentes de hidrogeno
El enlace puente de hidrógeno es una atracción que existe entre un átomo
de hidrógeno (carga positiva) con un átomo de O, N o X (halógeno) que posee
un par de electrones libres (carga negativa).
Esta complementariedad de pares de bases es consecuencia del tamaño, la forma y la
composición química de las bases. La presencia de miles de estos enlaces de hidrógeno en
una molécula de DNA contribuye en gran parte a la estabilidad de La doble hélice. Las
interacciones hidrófobas y de van de Waals entre los pares de bases adyacentes apilados
también contribuyen a la estabilidad de la estructura del DNA.
Las moléculas de azúcar se unen entre sí a través de grupos fosfato, que forman enlaces
fosfodiéster entre los átomos de carbono tercero (3′, «tres prima») y quinto (5′, «cinco
prima») de dos anillos adyacentes de azúcar. La formación de enlaces asimétricos implica
que cada hebra de ADN tenga una dirección. En una doble hélice, la dirección de
los nucleótidos en una hebra (3′ → 5′) es opuesta a la dirección en la otra hebra (5′ → 3′).
Esta organización de las hebras de ADN se denomina antiparalela; son cadenas paralelas,
pero con direcciones opuestas.
Almacenamiento de la información génica
Secuencias de DNA
integran
Genes
integran
Genoma, que es toda la información necesaria
Para la homeostasis de un organismo.

4
2. Replicación
Ciclo celular
El ciclo celular es un conjunto ordenado de sucesos que conducen al crecimiento de
la célula y la división en dos células hijas. El ciclo celular se inicia en el instante en que
aparece una nueva célula, descendiente de otra que se divide, y termina en el momento en
que dicha célula, por división subsiguiente, origina dos nuevas células hijas.
Interfase
Es el período comprendido entre divisiones celulares. Es la fase más larga del ciclo
celular, ocupando casi el 95% del ciclo, trascurre entre dos mitosis y comprende tres
etapas:
Fase G1 (del inglés Growth o Gap 1): Es la primera fase del ciclo celular, en la que
existe crecimiento celular con síntesis de proteínas y de ARN. Es el período que
trascurre entre el fin de una mitosis y el inicio de la síntesis de ADN. Tiene una duración
de entre 6 y 12 horas, y durante este tiempo la célula duplica su tamaño y masa debido
a la continua síntesis de todos sus componentes, como resultado de la expresión de
los genes que codifican las proteínas responsables de su fenotipo particular. En cuanto
a carga genética, en humanos (diploides) son 2n 2c.
Fase S (del inglés Synthesis): Es la segunda fase del ciclo, en la que se produce
la replicación o síntesis del ADN, como resultado cada cromosoma se duplica y queda
formado por dos cromátidas idénticas. Con la duplicación del ADN, el núcleo contiene el
doble de proteínas nucleares y de ADN que al principio. Tiene una duración de unos 6-8
horas.
Fase G2 (del inglés Growth o Gap 2): Es la tercera fase de crecimiento del ciclo celular
en la que continúa la síntesis de proteínas y ARN. Al final de este período se observan al
microscopio cambios en la estructura celular, que indican el principio de la división
celular. Tiene una duración entre 3 y 4 horas. Termina cuando la cromatina empieza a
condensarse al inicio de la mitosis. La carga genética de humanos es 2n 4c, ya que se
han duplicado los cromosomas, teniendo ahora dos cromátidas cada uno.
Replicación El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es
decir, sintetizar una copia idéntica). De esta manera de una molécula de ADN única, se
obtienen dos o más "clones" de la primera. El proceso de replicación es preciso, exacto y

5
monitoreado cuidadosamente porque un cambio en el DNA va a producir una información
incorrecta.
Retos :
Separar las dos bandas.
Dirección de la síntesis.
Proteger de errores durante el proceso.
La replicación es semiconservativa
Esta duplicación del material genético se produce de acuerdo con un mecanismo
semiconservador lo que indica que las dos cadenas complementarias del ADN original, al
separarse, sirven de molde cada una para la síntesis de una nueva cadena complementaria
de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas
del ADN original. Gracias a la complementariedad entre las bases que forman la secuencia
de cada una de las cadenas, el ADN tiene la importante propiedad de reproducirse
idénticamente, lo que permite que la información genética se transmita de
una célula madre a las células hijas y es la base de la herencia del material genético.
Proceso de replicación
El proceso está constituido
por 3 fases:
1. Iniciación
2. Elongación
3. Terminación
1. Iniciación La replicación comienza con el desenrrollamiento del DNA al romperse los puentes de
hidrogeno, se da en sitios específicos u orígenes de replicación, dependiendo de la célula
son los sitios u orígenes; en procariontes solo hay un origen de replicación en cambio en
eucariontes hay varios orígenes de replicación.
Procariontes, secuencias cortas consenso (conservadas) repetidas:
Secuencias palindrómicas (TATA)
Regiones ricas en A=T
Bacteriófago lambda (ori l; 80-120 pb)

6
Escherichia coli (ori C; 245 pb)
Eucariontes:
ARS=autonomously replication sequence
En Levaduras: 100-200 pb
En mamíferos: Múltiples segmentos de tamaño grande.
La dirección de la replicación puede ser:
• Unidireccional: mitocondria y virus
• Bidireccional: procariotas y eucariotas, es bidireccional a partir del origen de
replicación.
Horquilla de replicación
Es el sitio en donde la hélice de DNA se abre para comenzar la replicación, la helicasa es
la proteína desestabilizadora del DNA rompiendo puentes de hidrógeno para separar las
cadenas.
En E. coli el movimiento de la horquilla de replicación es de 1000 pb/seg, y se tarda
approx. 42 min en duplicar el genoma entero (4.5 x 10 6 pb)
En DNA eucariontes, la horquilla de replicación se mueve 100 pb/seg.
En humanos la replicación del DNA ( 109 pb) requiere approx 8 hrs, mientras que en
Drosophila toma de 3-4 min.
Topoisomerasas
Son enzimas que cortan y ligan el DNA cambiando su topología, por medio de giros o
relajando la molécula. Existen dos tipos: Topoisomerasa I y II.
Mecanismo: Cortan el DNA produciendo un enlace fosfodiester entre el grupo P y la
tirosina de la enzima utilizando magnesio:
Blanco de los agentes utilizados en quimioterapia:
La camptotecina ejerce acción antitumoral inhibiendo la topoisomerasa I
Las antraciclinas inhiben a la topoisomerasa II
Topoisomerasa I
Endonucleasa que interactúa con el DNA superenrollado
Corta una de las dos hebras, pasando la otra a través del corte, uniendo
nuevamente la hebra.
Provoca el relajamiento progresivo del DNA
No requiere de ATP para actuar
Topoisomerasa II
Endonucleasa que requiere ATP.
Interactúa con el DNA superenrollado negativamente.
Induce el corte y ligamiento de la doble hebra para relajar la molécula

7
Las girasas son topoisomerasas II
Eucariontes : TopII
Procariontes: girasa (E. coli)
Balero replicativo
Objetivo: Facilita el desplazamiento de la DNA polimerasa
Replicasa: adaptador del balero, balero y la enzima.
Funciones:
Evita que la DNA poli se salga de la hélice antes de concluir la síntesis
Regula el inicio y final de la síntesis
Tipos de baleros replicativos
Organismo Balero replicativo
Bacteriofago T4 gp45
E. coli Proteína β
Humano PCNA
DNA Polimerasa
Las DNA polimerasas intervienen en la replicación del ADN
para dar a cada célula hija una copia del ADN original en el
proceso de la mitosis. Cataliza la replicación de DNA:
reacción de polimerización.
Necesita:
- cadena molde
- Mg2+ - nucleótidos - terminal 3` libre ( primer)
Funciones principales de DNA polimerasa
- Síntesis
- Proofreading: actividad 3’ 5’
- Reparación: 5’ 3’
Tipos de enzimas:
- DNA Polimerasa I: repara y sintetiza.
- DNA Polimerasa III: enzima principal de la polimerización.
- DNA Polimerasa II, IV, y V: enzimas de reparación.
E. coli
- DNA pol I corrección durante la replicación, recombinación y

8
reparación por su actividad correctora de pruebas 5’ 3´. 16-20
nt/seg
- DNA pol II, mecanismos de reparación
- DNA pol III replicación del DNA > 500,000 nt/seg
- DNA pol IV y V reparación del DNA
mamífero
- Síntesis de hebra retrasada (lagging strand)
- Reparación de DNA
- Síntesis de DNA mitocondrial
- Síntesis de hebra líder (leading strand)
- Mecanismos de reparación de DNA
2. Elongación
Durante la elongación la ADN Pol III cataliza la síntesis de las nuevas cadenas añadiendo
nucleótidos sobre el molde. Esta síntesis se da bidireccionalmente desde cada origen, con
dos horquillas de replicación que avanzan en sentido opuesto. Cuando el avance de dos
horquillas adyacentes las lleva a encontrarse, es decir, cuando dos burbujas se tocan, se
fusionan, y cuando todas se han fusionado todo el cromosoma ha quedado replicado.
Hebra líder (leading strand) al mismo sentido que la horquilla de replicación.
• Primasa
• DNA pol III
• DNA pol I sustituye el RNA primer por nucleótidos
Puesto que la holoenzima ADN Pol III necesita de un extremo 3'-OH libre, es necesario
que una ARN primasa catalice la formación de un fragmento corto específico
de ARN llamado cebador, que determinará el punto por donde la ADN polimerasa
comienza a añadir nucleótidos. Así, durante la síntesis, en cada horquilla de replicación se
van formando dos copias nuevas a partir del cebador sintetizado en cada una de las dos
hebras de ADN que se separaron en la fase de iniciación, pero debido a la unidireccionalidad
de la actividad polimerasa de la ADN Pol III, que sólo es capaz de sintetizar en sentido 5´
→ 3', la replicación sólo puede ser continua en la hebra adelantada; en la hebra rezagada es
discontinua, dando lugar a los fragmentos de Okazaki.
En la hebra rezagada, cuando la ADN Pol III hace contacto con el extremo de otro
fragmento de Okazaki contiguo, el cebador de ARN de éste es eliminado y los dos
fragmentos de Okazaki de ADN recién sintetizado son unidos. Una vez todos juntos se
completa la doble hélice de ADN. La eliminación de cebadores también se da en la hebra
conductora, de síntesis continua, pero debido a que en ésta hay un solo cebador es un
proceso que sólo tiene lugar una vez, mientras que en la hebra rezagada se dará tantas

9
veces como fragmentos de Okazaki haya. Para ello intervienen una serie de enzimas: la
enzima RNasa H ("H" de híbrido ARN-ADN) elimina el cebador a excepción del
ribonucleótido directamente unido al ADN; la ADN Pol I elimina este ribonucleótido gracias
a su actividad exonucleasa 5' → 3' y rellena el hueco con ADN quedando una molécula
completa a excepción de una rotura (o "mella") entre el extremo 3'-OH libre y el fosfato 5'
de la cadena reparada; por último, la ADN ligasa sella esa rotura catalizando la reacción de
condensación entre el grupo fosfato y el OH de la desoxirribosa del nucleótido contiguo
completando el enlace fosfodiéster.
Hebra retrasada (lagging strand) al sentido opuesto de la horquilla de replicación.
• Formación de una asa
• Primasa
• DNA pol III
• DNA pol I
• DNA ligasa sella los enlaces.
3. Terminación
Las horquillas de replicación llegan a una secuencia de 20 pb denominada Ter a la cual se
une una proteína de terminación (Tus) y se separan los complejos y las hebras sintetizadas.
Telomeros
Los telómeros son secuencias cortas de 5-8 pb, ricas en G, repetidas en tandem,
localizadas al final de los extremos de los cromosomas lineales de la mayoría de los
eucariotas.
Son secuencias no codificantes
Son necesarios para el mantenimiento y la estabilidad de los cromosomas.
Los telómeros tienden a acortarse en cada ronda de replicación en la hebra rezagada,
porque :
• DNApolimerasa no puede actuar sin primer
• Primasa no puede colocar un primer , si no tiene un molde de DNA
• La hebra conductora se replica por completo sin interrupción
Telomerasa
Compuesta de un componente proteico (subunidad catalítica hTERT) y una molécula
de RNA
Es una Ribonucleoproteína con actividad DNA polimerasa.
Añade secuencias teloméricas en el extremo 3’ del DNA (~transcriptasa inversa)
La molécula de RNA de la Telomerasa sirve de molde para añadir repeticiones de 6 nt
al extremo 3’ de los telómeros.
Este alargamiento permite añadir el primer para la DNApol
La telomerasa se activa en muchas células tumorales

10
Papel en el envejecimiento(“reloj biológico”), la proliferación y el cáncer
Reparación de DNA
El mantenimiento de la información contenida en el DNA es fundamental, el DNA es
irreemplazable (a diferencia del RNA o las proteínas). Puede resultar dañado por diversos
procesos (espontáneos, agentes, ambientales)
Cuando estas modificaciones no se reparan después de varios ciclos de replicación, son
irreversibles y repercuten en la función y estructura de los organismos, la replicación puede
dejar bases mal apareadas.
DNA es la única molécula que es reparada en lugar de ser degradada y la eficacia del
proceso de 1000 en 1, sólo uno de cada mil errores se repara.
Dos procesos de chequeo:
Generado por la polimerasa
Prueba de lectura exonucleolitica en dirección 3’ 5’
El proceso está compuesto de tres partes:
La porción alterada es reconocida y eliminada por medio de las nucleasas de
reparación. Produciendo un hueco.
DNA poli une los nucleótidos necesarios
DNA ligasa une las bases