cicy.repositorioinstitucional.mx · Figura 3.2. Secuencia completa del gen KASII y diseño de...
Transcript of cicy.repositorioinstitucional.mx · Figura 3.2. Secuencia completa del gen KASII y diseño de...





Este trabajo se llevó a cabo en la Unidad de Bioquímica y Biología Molecular de Plantas
del Centro de Investigación Científica de Yucatán A C, y forma parte del proyecto titulado
MEJORAMIENTO GENÉTICO DE Jatropha curcas MEDIANTE TÉCNICAS
BIOTECNOLÓGICAS con apoyo de la BIORED bajo la dirección delDr. Víctor Manuel
Loyola .

AGRADECIMIENTOS
En primer lugar agradezco infinitamente a DIOS, por que sin su fortaleza y
sabiduría, no hubiera sido posible levantarme de duros golpes, porque me enseñó con
su amor a luchar y valorar las dos cosas más preciadas: la vida y a la familia.
Al CONACYT por la beca No. 265339 otorgada durante el tiempo de esta
formación.
Al Dr. Víctor M. Loyola Vargas¸ por haberme permitido formar parte de su grupo
de alumnos, por brindarme su apoyo académico durante el tiempo que permanecí en
su laboratorio desde la licenciatura.
A la MC. Rosa M. Galaz Avalos, por su dedicación hacia cada uno de los
experimentos que poníamos en marcha, por enseñarme las técnicas necesarias, pero
sobre todo por su apoyo incondicional desde que llegué a la ciudad, y por haberme
brindado una valiosa amistad.
A mi PADRE: por enseñarme a luchar hasta el último minuto de vida, por aferrarte
a mis manos el día en que partiste y motivaste a seguir en PIE y a no DECAER a
caminar siempre hacia la cima, GRACIAS PAPÁ por tu amor, por tu entereza y
valentía. A mi MADRE: por tantos sacrificios para darme la mejor educación, por
cada noche en vela, porque sacrificaste comodidades, por incomodidades, por llevarme
de la mano y decirme una y otra vez tu puedes, no decaigas, sigue, animo, todo
eso me permitió concluir esta etapa más.
A mis compañeros y amigos de LAB-24. Laura Rodríguez, Cristy Catzin, Heydi,
Marisa Morales, Sara Hernández, Yuri Espinosa, Celia Uc, Ruth Márquez, Ariana
Pérez, Pedro Xool, Filiberto Collí, Miguel Uc, Geovany Nic, Jorge García, Caleb
Reyes, Hugo Méndez, Randy Aviléz, Benjamín Ayil, Jacobo Pérez; Asi También De
La UBBMP: Yusira Cab, DINORA Canseco, Norma Mukul, Jony Avilez, Yesenia Díaz
Jorge Xool, De La Unidad De Materiales: Joel García, Anahy, Biotecnología: y
Energía Renovable: Nancy, Denisse y Gonzalo, por todos los momentos compartidos,

desvelos y diversiones que disfrutamos en esta etapa pero sobre todo por
permitirme conocerlos y regalarme su bella y valiosa amistad.
Así también a mis profesores, los cuales sin su ayuda no hubiera sido posible
culminar esta etapa y permitir formarme como MC.
Agradezco de manera especial a: MC. Gonzalo Herrera, Dr. José Walner Cadenas
Acuña, Dr. Arquímedes Oramas Vargas y Lic. José Del C. Herrera, por su apoyo
para los determinados gastos de titulación.

DEDICATORIAS
Este trabajo está realizado por el esfuerzo y entrega de muchos años, y en especial
a: A MIS PADRES: José Manuel Cadenas Acosta+ lo dedico a tu memoria, lucha y
valentía por enfrentarte a la VIDA, porque después de la MUERTE me has
demostrado que siempre estás conmigo, protegiendo de peligros y ayudando a no
desfallecer ante adversidades, te amo y extraño en cada momento; Ma. Teresa
González Pérez, eres mi mayor tesoro y por quien me he esforzado desde que
perdimos a nuestro pilar, por ti MADRE he llegado a donde estoy y que hemos
pasado adversidades, caídas, malos momentos los cuales se borran al ver tu sonrisa y
mirada cada DIA, TE AMO MADRE.
A MI ABUELA.DORA ACOSTA DE LOS SANTOS, por ser mi GRANDMOTHER y
enseñarme a que en esta vida los tropiezos y caídas no importa siempre y cuando
te sepas Levantar con el pie derecho, me has enseñado a saber vivir con FÉ
A MI GRAN FAMILIA (CADENAS Y GONZÁLEZ) por la distancia, de los años,
donde ocasioné las preocupaciones y nostalgias, pero muy por encima de todo a base
de SACRIFICIOS pudimos lograr esto LOS ADORO.
A MIS AMIGAS(OS) de años de locuras, que con cada uno he compartido y me han
dado todo su apoyo y confianza para formar parte de mi vida, por aconsejarme en
el momento justo: Sandra Barrosa, Isa Almeida, Xochitl Alejo, Josefina Adriano,
Karen Sánchez, Tomasa Montero, Dorothy Rosique, Jenifer Vidal, Lupita Torres;
Walner Cadenas, Iván Ramos, Freddy Cruz Y Cristian Candelero, porque la distancia
no importa para llevar una amistad.sincera.desde muy lejos.

i
ÍNDICE
INTRODUCCIÓN .................................................................................................................... 5
CAPÍTULO I ........................................................................................................................... 6
ANTECEDENTES ............................................................................................................................. 6
1.1. JATROPHA CURCAS ................................................................................................................ 10
1.1.1. CLASIFICACIÓN TAXONÓMICA ................................................................................................. 11
1.1.2. MORFOLOGÍA VEGETAL ........................................................................................................... 11
1.1.3. IMPORTANCIA DEL CULTIVO ..................................................................................................... 13
1.1.4. COMPONENTES QUÍMICOS ...................................................................................................... 15
1.1.5. AMBIENTE O LOCALIDAD .......................................................................................................... 15
1.2. BIOSÍNTESIS DE LÍPIDOS EN PLANTAS ........................................................................................ 16
1.2.2. ÁCIDO GRASO SINTASA. .......................................................................................................... 21
HIPÓTESIS ...................................................................................................................................... 30
OBJETIVO GENERAL .................................................................................................................... 30
OBJETIVOS ESPECÍFICOS .......................................................................................................... 30
JUSTIFICACIÓN ............................................................................................................................. 30
CAPÍTULO II ........................................................................................................................ 33
MATERIALES Y MÉTODOS .......................................................................................................... 33
ESTRATEGIA EXPERIMENTAL ............................................................................................................. 33
........................................................................................................................................................... 34
2.1. INTRODUCCIÓN ..................................................................................................................... 35
2.2.1. ALINEAMIENTO DE SECUENCIAS DE AMINOÁCIDOS ................................................................. 36
2.2.2. DISEÑO DE OLIGONUCLEÓTIDOS ............................................................................................. 36
2.2.3. MATERIAL VEGETAL DE J. CURCAS ......................................................................................... 36
2.2.4. EXTRACCIÓN DE ARN. ............................................................................................................ 37

ii
2.2.5. SÍNTESIS DE CDNA POR RT-PCR ......................................................................................... 38
2.2.6. CONDICIONES DE PCR ........................................................................................................... 39
2.2.7. ANÁLISIS DE EXPRESIÓN DE JCKASII MEDIANTE QPCR........................................................ 40
2.2.8. TRANSFORMACIÓN DE LA CEPA E. COLI DH5-Α ..................................................................... 41
2.2.9. PROTOCOLO DE MINIPREPARACIÓN DE ADN PLASMÍDICO {BIRNBOIM, 1997 31478 /ID} ..... 42
2.3. MAPA DE LOS PLÁSMIDOS PCAMBIA2301, PGEM®-T EASY .................................................. 43
2.3.1. PROTOCOLO PARA OBTENCIÓN DE CÉLULAS COMPETENTES ................................................. 44
2.3.2. PREPARACIÓN DEL CULTIVO DE A. TUMEFACIENS .................................................................. 45
2.3.3. PREPARACIÓN DE CÉLULAS COMPETENTES DE A. TUMEFACIENS .......................................... 46
2.3.4. TRANSFORMACIÓN DE LA CEPA BACTERIANA A. TUMEFACIENS ............................................. 46
2.3.5. TRANSFORMACIÓN MEDIANTE AGROBACTERIUM EN TEJIDOS IN VITRO DE J. CURCAS ......... 47
2.3.6. LOCALIZACIÓN HISTOQUÍMICA DE GUS EN TEJIDOS IN VITRO DE JATROPHA ........................ 47
2.4. OBTENCIÓN DEL MATERIAL VEGETAL IN VITRO........................................................................... 48
2.4.1. INDUCCIÓN DE CALLO .............................................................................................................. 50
CAPÍTULO III ....................................................................................................................... 54
RESULTADOS Y DISCUSIÓN ...................................................................................................... 54
3.1. INTRODUCCIÓN ..................................................................................................................... 54
3.2. ALINEAMIENTO DE KASII. .......................................................................................................... 54
3.2.1. DISEÑO DE OLIGONUCLEÓTIDOS ............................................................................................. 56
3.3.1. ANÁLISIS DE EXPRESIÓN DE KASII MEDIANTE QPCR ............................................................ 60
3.3.2. PATRÓN DE EXPRESIÓN DEL GEN KASII EN DIFERENTES DÍAS POST-ANTESIS DE FRUTOS .. 61
3.3. CLONACIÓN DEL GEN JCKASII EN EL VECTOR PGEM ®- T EASY ....................................... 69
3.3.1. ANÁLISIS DE DE LA SECUENCIACIÓN ....................................................................................... 73
3.4.1. EFECTO DE LOS ANTIBIÓTICOS EN EL CRECIMIENTO/RESISTENCIA DE LA BACTERIA ............. 77
3.5.1. INDUCCIÓN DE CALLO .............................................................................................................. 80
3.5.2. EFECTO DE LOS ANTIBIÓTICOS SOBRE EL MATERIAL NO TRANSFORMADO ............................ 85

iii
3.6. DESARROLLO DE PROTOCOLO DE TRANSFORMACIÓN DE CALLOS DE J. CURCAS CON
PCAMBIA2301 ................................................................................................................................. 88
CAPÍTULO IV ....................................................................................................................... 93
DISCUSIÓN GENERAL .................................................................................................................. 93
4.1. CONCLUSIONES ................................................................................................................ 94
4.2. PERSPECTIVAS. .................................................................................................................... 94
REFERENCIAS BIBLIOGRÁFICAS ............................................................................................. 96

iv
LISTADO DE FIGURAS
Figura 1.1. Distribución de los costos de producción de biodiésel a partir de J. curcas [6]. .............. 9
Figura 1.2. Mapa geográfico de Jatropha en México. ....................................................................... 10
Figura 1.3. Partes importantes de la planta. a) Floración en la rama, b) corteza, c) limbo de la hoja,
d) pistilo de la flor, e) flores, f) corte transversal de la fruta inmadura, g) frutas, h) corte longitudinal
de los frutos, i) semilla [10]. ............................................................................................................... 12
Figura 1.4 A. Semillas. B. Frutos inmaduros de J. curcas. ............................................................... 13
Figura 1.5. A) Inflorescencia de J. curcas que contiene tanto flores masculinas como femeninas; B.
Arbusto; C. Semillas maduras y fruto abierto; D) Semillas maduras de J. curcas; E. Hojas foliares.
........................................................................................................................................................... 14
Figura 1.6. Condiciones climáticas más favorables para el cultivo de J. curcas y de la palma de
aceite. ................................................................................................................................................ 16
Figura 1.7. Síntesis de los principales ácidos grasos saturados, insaturados y acil-CoA en las
células vegetales. Los grupos acilo de ácido graso son nombrados por el número de carbonos:
número de dobles enlaces 2:00: acetilo; 3:00: malonil; 4:0: butiril; 16:00: palmitoil (hexadecanoato);
18:0: esteárico (octadecanoato); 18:1: oleoil (octadecenoato); ACP: proteína acil portador; ACS:
acil-CoA-sintetasa; KAS: 3-cetoacil-ACP sintasa, TE: tioesterasa. .................................................. 18
Figura 1.8. Precursores de la síntesis de los ácidos grasos. ............................................................ 19
Figura 1.9. Ruta de biosíntesis de los ácidos grasos en Jatropha. Los íconos al lado de cada
nombre de la enzima muestran el número total de ESTs correspondientes a esa enzima en JD y
JG, por separado. KAS I participa desde el 2º al 6º ciclo de extensión KAS II participa en el 7º ciclo
de extensión de la cadena de carbonos. Ambos FatA y FatB pueden catalizar la conversión de
18:0-ACP en ácido esteárico. Los ácidos grasos producidos por esta vía son 16:0 (ácido palmítico),
18:0 (ácido esteárico), 18:1 (ácido oleico) y 18:2 (ácido linoleico) [23]. ........................................... 21
Figura 2.1. Estrategia experimental. ................................................................................................. 34
Figura 2.4. Material vegetal de Jatropha curcas de A. Plantula in vitro. B. Frutos inmaduros. C.
Fruto con Semillas maduras. ............................................................................................................. 37
Figura 2.3.Condiciones de amplificación para los diferentes pares de oligonucleoticos utilizados. . 40
Figura 2.5. Mapa de restricción de pCAMBIA2301. Se observa el tamaño del plásmido (11, 633 pb),
el promotor constitutivo CAMV35S. Sitios de restricción de enzimas, gen de resistencia para
selección bacteriana, HYG (higromicina) y gen de resistencia para selección de plantas
transformadas Kanamicina. ............................................................................................................... 43

v
Figura 2.6. Vector de clonación pGEM®-T Easy. Se observa el tamaño (3016 pb), el promotor T7,
Sitios de restricción de enzimas, gen de resistencia para selección bacteriana, AmpR (ampicilicina).
........................................................................................................................................................... 44
Figura 2.7. Protocolo de inducción para J. curcas ............................................................................ 51
Figura 3.1. Alineamiento de secuencias para KASII de J. curcas con proteínas de otras especies.
Los aminoácidos marcados en el mismo color indican la identidad. El sitio activo Cys324 se indica
con flecha vertical color azul. ............................................................................................................ 56
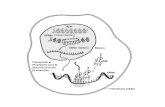
Figura 3.2. Secuencia completa del gen KASII y diseño de oligonucleótidos. Las flechas indican el
plegamiento de los cebadores diseñados (5´ - 3´). ........................................................................... 57
Figura 3.3. Gel de agarosa con muestras de ARN en diferentes tejidos vegetales; H. Hojas, T tallo,
Semilla en diferentes días post-antesis (dpa). .................................................................................. 59
Figura 3.4. Analisis dee la expresion cuantitativa de KASII en los diferentes dias post-antesis. El
nivel de expresion fue mayor a los 30 dpa. Los niveles de expresion fueron estandarizados con la
JcActina, para cada muestra se utilizaron tres replicas. ................................................................... 61
Figura 3.5. Amplificación del transcrito ActinaJc con 230 pb de J. curcas, a partir de 3 µg de ARN.
Se tomó 1 µL de ADNc. Se cargó 10 µL de producto de PCR. Gel de agarosa al 1 % y teñido con
Gel red. T. Tallo, H: Hoja, Días post-antesis. .................................................................................... 61
Figura 3.6. Gel de agarosa en donde se observa la expresión de JcKASII en semillas de maduras
(30 dpa) obtenidos con los cebadores PAR2 de un tamaño de 239 pb. .......................................... 62
Figura 3.7. Amplificación del transcrito de JcKASII con los cebadores PAR3, obtenido a los 35 dpa.
Se cargó 10 µL de producto de PCR. Gel de agarosa al 1 % y teñido con Gel red. MM: Marcador
de 1 Kb. 1: 72, 2:71.1, 3: 67.3, 4:64.4, 5:62.3 y 6:60°C. ................................................................... 62
Figura 3.8. Expresión de JcKASII con cebadores del PAR4 aprox. con 1312 pb. Se observan
bandas inespecíficas en los carriles a diferentes gradientes de temperatura, se pudo amplificar un
tamaño aprox de 1000 y de 1300 pb. ............................................................................................... 63
Figura 3.9. Gel de agarosa donde se puede observar la amplificación del transcrito de JcKASII
generados con PAR 4, a los 25, 30 y 35 dpa. Se cargó 10 µL de producto de PCR. Gel de agarosa
al 1 % y teñido con Gel red. MM: Marcador de 1 Kb. ....................................................................... 64
Figura 3.10. A. Combinación de cebadores C1 (R/F) PAR2 Y PAR 3; CASO 2 (F/R) PAR 3 Y PAR
2, A 50, 55 Y 60°C Día 25 post-antesis. B. Combinación de cebadores C1 (R/F) PAR2 Y PAR 3;
CASO 2 (F/R) PAR 3 Y PAR 2, A 50, 55 Y 60°C Día 30 post-antesis; se utilizó ActinaJc como
testigo. ............................................................................................................................................... 64

vi
Figura 3.11. Expresión de JcKASII con cebadores del PAR3 y PAR 4 con un tamaño esperado de
1722 y 1312 pb respectivamente. Se observan bandas inespecíficas en los carriles a diferentes
gradientes de temperatura con un tamaño aprox de 1000 y 1600 pb para PAR3 y de 1300 pb para
el PAR 4. Se utilizó como gen constitutivo ActinaJc con un tamaño de 230 pb. .............................. 65
Figura 3.12. Gel de agarosa donde se observa la expresión de JcKASII utilizando cebadores del
PAR5 aprox. con 1700 pb, bandas inespecíficas debido a los gradientes de temperatura,
amplificando un tamaño de 1000 pb. Se usaron testigos positivos como PAR 2 (JcKASII de 289 pb)
y PAR1 (ActinaJc de 200 pb) y el testigo negativo. .......................................................................... 66
Figura 3.13. Amplificación del transcrito de KASII de J. curcas a los 30 dpa. Se cargó 10 µL de
producto de PCR. Gel de agarosa al 1 % y teñido con Gel red. MM: Marcador de 1 Kb. 1: 50, 2:52,
3: 55.3, 4:55.6, 5:60 y 6:60.8 °C. ...................................................................................................... 67
Figura 3.14. Gel de agarosa de la expresion del ADNc del gen JcKASII con los cebadores
especificos (PAR3) de un tamaño de 1722 pb. La reacción fue llevada a cabo con el ADNc del dia
25 dpa, con condiciones de amplificacion descritas en el manual de la Taq y utilizando 3
temperaturas de alineamiento; además de testigos (PAR 1 ActinaJc y KASIIJc de 289 pb). .......... 68
Figura 3.15. Producto de PCR purificado a diferentes diluciones de muestra. ................................ 70
Figura 3.16. Gel de Agarosa donde se observa la integridad del ADN plasmídico de las clonas
seleccionadas. Se cargó 2 µL de ADNg en el gel teñido con Red Gel ®. Los números azules
indican el número correspondiente a cada clona. ............................................................................. 72
Figura 3.17. Electroforesis en gel del resultado de la PCR realzada a las colonias seleccionadas
usando cebadores específicos PAR3 y PAR5. Se observa el fragmento esperado (1722 pb) para
las clonas de la 6 a la 10 y de la 12 a la 16. Los números rojos indican el numero de clona
correspondiente. ................................................................................................................................ 72
Figura 3.18. Alineamiento de secuencias generadas para JcKASII. Las flechas rojas Derecha a
izquierda (5´a 3´) indican la secuencia que obtuvimos, ademas del empalmamiento de los
oligonucleotidos y la enzima de restricción HindIII; Flecha negra corresponde al fragmento
completo para KASII reportado en el GenBank de 1722 pb. ............................................................ 74
Figura 3.19. Alineamiento de la secuencia correspondiente al ADNc de JcKASII comparada con la
depositada en el Gen Bank. .............................................................................................................. 75
Figura 3.20. Cultivo y activación de cepas de A. tumefaciens. ......................................................... 77
Figura 3.21. Preparación de antibióticos y medio YEB para bacteria............................................... 78

vii
Figura 3.22. Ciclo de obtención de embriones cigóticos de J. curcas.1. Obtencion de frutos y lavado
de semillas; 2. Desinfección en condiciones de asepxia; 3. Extracion del embrion cigótico y 4.
Germinacion en medio MS. ............................................................................................................... 79
Figura 3. 23. Proceso general del desarrollo de los embriones para obtención de las plántulas in
vitro. A. Germinación de embrion cigótico; B. Día 2. C y D. Día 4 Enloganción de tallo. E-G. Día 7.
Abertura de los cotiledones. H-I. Día 10. Curvatura del tallo. ........................................................... 80
Figura 3.24. Formación de callos y brotes in vitro de J. curcas con tratamiento de BA+ IBA 2.22 Y
4.92 µM) obtenidos de diferentes explantes. A. Hipocotilo; B. Hoja; C. Hoja cotiledonar; D. Plántula
completa parte radical. ...................................................................................................................... 81
Figura 3.25. Formación de callos y brotes in vitro de J. curcas con tratamiento de BA+ IBA+KIN
(4.44, 1.23 Y 2.12 µM) obtenidos de diferentes explantes. A. Hipocotilo; B. Hoja; C. Hoja
cotiledonar; D. Plántula completa parte radical. ................................................................................ 82
Figura 3.26. Embriogénesis somática de J. curcas con tratamiento BA (2.92 µM) y de AIA (0.57 µM)
(MJC2) obtenidos a partir de callo. .................................................................................................... 82
Figura 3.27. Embriones somáticos en diferentes estadios de desarrollo se observan estadios muy
definidos y otros con algunas deformaciones. .................................................................................. 83
Figura 3.28. Respuesta embriogénica de embriones por matraz del tratamiento MJCT2. ............... 84
Figura 3.29. Regeneración de brotes de J. curcas y obtención de organogénesis. ......................... 84
Figura 3.30.Brote embriogénico con 3 meses de crecimiento, se observan algunas esstructura con
forma de cotiledones. ........................................................................................................................ 85
Figura 3.31 Efecto de la Kanamicina suministrados a callos de J. curcas en diferentes
concentraciones. Callos en el Día 0 de tratamiento se muestran los callos bien regenerados y sin
daño alguno; Segundo panel. En el centro de cada caja petri con medio MS, se muestra al testigo
(sin antibiotico) y alrededor de el al expueso durante 30 dias, obserbandose un cambio en su
fisiología. ........................................................................................................................................... 86
Figura 3.32. Efecto de la Higromicina suministrados a callos de J. curcas en diferentes
concentraciones. Primer panel. Callos en el Día 0 de tratamiento se muestran los callos sin daño
alguno; Segundo panel. En el centro de cada caja petri, se muestra al testigo y alrededor de el al
tejido expuesto al antibiótico después de 30 días con las concentraciones 2.5 y 10 mg/L la dosis es
letal. ................................................................................................................................................... 87
Figura 3.33.Efecto de Cefotaxima (400 mg/L) suministrado a callos de J. curcas a los 30 días se
puede observar que los callos no tienen daño celular abundante. ................................................... 88

viii
Figura 3.34. Localización histoquímica de GUS en callos no embriogénicos de Jatropha sin
infiltración al vacio utilizando una concentración de 40 mg X-Gluc por mL de DMSO. A. Callo en
estadio de desarrollo. B. Acercamiento de tejido teñido. C. callo con malformación a 10x y D.
acercamiento de tejido a 40x. ........................................................................................................... 89
Figura 3.35. Tinción histoquímica de GUS en diferentes tejidos vegetales de J. curcas. A. Callo
verde y blanco, en la primera columna se observa el testigo y en las siguientes los tejidos que
fueron sometidos a la tinción; B. Estructuras embriogénicos en estados de desarrollo; C. Embrión
cigótico utilizando un testigo y los transformados teñidos con el gen reportero uidA. ..................... 90
Figura 3.36. Dosis respuesta del X-Gluc en cultivos in vitro de Jatropha curcas transformados con
la ceba LBA4404::pCAMBIA2301. A y B. Embriones somáticos utilizando una concentración de
0.02 M (1 mg/mL) de X-Gluc; C y D. Callo verde teñido a una concentración de 0.02 M; E y F.
Embriones somáticos tenidos con X-Gluc a una concentración de 1.8 M (40 mg/mL). El sustrato 5-
bromo-4-cloro-3-indolyl-β-glucorinida fue disuelto con 1 mL de Dimetilsulfoxido utilizando un pH de
amortiguador de sustrato de 7. ......................................................................................................... 91
Figura 3.37. Protocolo general de transformación de callos embriogénicos de J. curcas. A. Plántula
a 15 días de germinación medio liquido; B. Formación de callo en el tallo de la plántula; C.
Estructuras originadas después de 60 días de inducción (BA+AIA) medio liquido; D. Organogénesis
obtenida a 30 días después de inducción; E. Estructuras embriogénicas con algunas
malformaciones; F. Callo de 2 meses de desarrollo; G. Callo embriogénico transformado con el gen
uidA tratamiento 1 mg/Ml; H-I. Embrión transformado después de 1 mes de teñido. ...................... 91

ix
LISTADO DE TABLAS
Tabla 1.1. Contenido de aceite y composición de ácidos grasos de varias oleaginosas [5]. ............. 8
Tabla 1.2. Producción de aceite de origen vegetal [5]. ....................................................................... 8
Tabla 1.3. Composición química del aceite de Jatropha. ................................................................. 15
Tabla 1.4. Enzimas involucradas en la biosíntesis de los ácidos grasos que forman el aceite en las
semillas de J. curcas. ........................................................................................................................ 20
Tabla 1.5. Complejo Proteico de la ácido graso sintasa. .................................................................. 23
Tabla 1.7. Condiciones óptimas para la esterificación del aceite de J. curcas. ................................ 25
Tabla 1.8. Resumen general de trabajos reportados en transformación de J. curcas ..................... 29
Tabla 2.1. Componentes del amortiguador de X-GlcA ..................................................................... 48
Tabla 2.2. Composición de medio MS para germinación. ................................................................ 50
Tabla 2.3. Medio de inducción de callos (IC) .................................................................................... 51
Tabla 2.4. Medio de inducción de brotes (IB). .................................................................................. 52
Tabla 3.1. Números de accesión de las secuencias de aminoácidos de las KASII seleccionadas
para este estudio. .............................................................................................................................. 54
Tabla 3.2. Genes involucrados en la síntesis de ácidos grasos. ...................................................... 55
Tabla 3.3. Secuencias de oligonucleótidos y longitud de amplicones correspondientes (5´-3´). ..... 58
Tabla 3.4. Volumen de reacción utilizada en la ligación. .................................................................. 70
Tabla 3.5. Concentracion de ADN genómico en las clonas que se obtuvieron ................................ 71
Tabla 3.6. Porcentaje de identidad comparado con otras especies de la secuencia obtenida ........ 73
Tabla 3.7. Listado de antibióticos utilizados en la transformación. ................................................... 77
Tabla 3.8. Antibióticos en diferentes combinaciones para de A. tumefaciens. ................................. 78

x
ABREVIATURAS
Ácido indol acético AIA
Benciladenina BA
Ácido indol butírico IBA
Medio de Murashige y Skoog MS
Medio de Gamborg B5
Regulador del crecimiento vegetal RCV
Plant Preservative Mixture PPM
Ácido graso sintasa KAS
Esteroil-ACP desaturasa SAD
Ácido desoxirribonucleico ADN
Ácido ribonucleico ARN
Dietilpolicarbonato DEPC
Reacción en cadena de la polimerasa PCR
ß-glucoronidasa GUS
Revoluciones por minuto rpm
Pares de base pb
Días post-antesis dpa
Ampicilina Amp
Cefotaxima Cf
Kanamicina Kan

xi

1
RESUMEN
La necesidad de desarrollar alternativas energéticas se ha acelerado debido a la
crisis energética mundial y a los problemas ambientales que genera el uso de varios
energéticos que se utilizan actualmente. Entre las fuentes renovables de energía que más
posibilidades tienen de reemplazar al petróleo se encuentran las energías eólica, solar y
nuclear, biomasa, entre otras. Los biocombustibles, representan el producto final de la
utilización de cultivos como la caña de azúcar, el maíz, la soja, el girasol, la colza y otros,
como Jatropha curcas.
En las últimas décadas, se ha descubierto que los frutos de Jatropha contienen
cantidades significativas de un combustible de aceite vegetal, que presenta características
ideales para la combustión y por lo tanto, se puede utilizar para la producción de energía
renovable y biocombustibles de 2ª generación. Por estas razones, miles de iniciativas
para el cultivo de Jatropha se han iniciado por las cooperativas de agricultores,
empresarios individuales, empresas y gobiernos de todo el mundo.
El conocimiento total de los genes involucrados en la ruta biosintética de los ácidos
grasos podría combinarse con la biología sintética y aprovechar algunos microorganismos
para producir bioenergía masivamente. En el caso de J. curcas, el uso de estos temas,
podría utilizarse producir una mayor cantidad de semillas, y de esta forma obtener un
aumento en la producción del aceite y por ende una mejora en la calidad del biodiésel.
En el presente trabajo, se propuso estudiar uno de los principales genes
involucrados en la biosíntesis de ácidos grasos y el desarrollo de un método para
transformar tejidos de J. curcas mediante la bacteria A. tumefaciens con un gen reportero
uidA. Una vez establecido el protocolo, se pretende utilizar en un futuro, mediante la
inserción de este gen que codifica para la β-cetoacil-ACP sintasa, y con ello se pretende
aumentar el contenido de ácido esteárico y disminuir el contenido de ácido palmitoleico, lo
que permitiría obtener un aceite más adecuado para la producción de biodiésel.

3
ABSTRACT
The need to develop alternative energy sources has accelerated due to the global energy
crisis and environmental problems caused by the use of various energy currently used.
Among the renewable energy sources that most likely to replace oil are the wind, solar,
nuclear, biomass, among others. Biofuels, considered as an intermediate step to the
development of renewable and safe energy, represent the final product of using crops
such as sugarcane, corn, soybean, sunflower, rapeseed and others, like Jatropha curcas.
In the past decades, it has been discovered that the fruits of Jatropha contain significant
amounts of vegetable oil fuel, which has ideal characteristics for combustion, and
therefore, can be used for the production of renewable and biofuels 2nd generation. For
these reasons, thousands of initiatives for growing Jatropha have been initiated by farmer
cooperatives, individual entrepreneurs, businesses and governments around the world.
The total knowledge of the genes involved in the biosynthetic pathway of fatty acids could
be combined with synthetic biology and take advantage of some microorganisms for
bioenergy massively. In the case of J. curcas using these issues could be used to produce
a greater number of seeds, and thus obtain an increased oil production and therefore an
improvement in the quality of the biodiesel.
In this paper, we propose to study one of the main genes involved in fatty acid
biosynthesis and a method for transforming tissues J. curcas by bacteria A. tumefaciens
with a reporter gene uidA. Once the protocol is to be used in the future, by inserting the
gene encoding β-ketoacyl-ACP synthase, this amendment is to increase stearic acid
content and decrease the content of palmitoleic acid, which that would yield an oil more
suitable for biodiesel production.
.

4

5
INTRODUCCIÓN
Actualmente, se busca encontrar alternativas energéticas que permitan reemplazar
los combustibles fósiles y de esta forma poder disminuir el impacto ambiental que éstos
causan, así como enfrentar la elevada demanda energética mundial y agotamiento de las
reservas petrolíferas, todo ello ha llevado a un cambio en el paradigma de la producción
de energía y generado un interés del uso de energías limpias o renovables [1].
Una alternativa, es la planta oleaginosa Jatropha curcas, la cual potencialmente,
puede ser usada en áreas deforestadas, erosionadas y en suelos marginales. Esta planta
es un miembro de la familia de las Euphorbiaceae que se localiza en climas tropicales y
semitropicales, es originaria de Mesoamérica, y se le puede cultivar en todas las zonas
tropicales del mundo.
De las semillas de J. curcas puede extraerse alrededor de un 40% de aceite
susceptible de ser procesado y transformado en biodiésel [2]. El aceite de Jatropha
contiene predominantemente ácidos linoleico y oleico, los cuales constituyen del 72 al
80% de los ácidos grasos totales. Otros ácidos grasos presentes en el aceite incluyen los
ácidos palmítico y esteárico, así como los ácidos caprílico, mirístico, palmitoleico,
araquídico, behénico y lignocérico [3].
El valor comercial del biodiésel está determinado por su índice de cetano. A su
vez, el índice de cetano está determinado por el grado de insaturación en los ácidos
grasos presentes en el aceite, así como por el número de ramificaciones. Por ello, una
alternativa importante para mejorar la calidad del aceite es disminuir la cantidad de
insaturaciones presentes en los ácidos grados del aceite de Jatropha. En este proyecto,
se analizó la expresión de KASII en frutos y tejido vegetal de J. curcas. Los resultados
permitieron identificar la expresión del gen KASII en hoja, tallo y fruto maduro e inmaduro,
posteriormente se obtuvo el ADNc de KASII completo para poder utilizarlo en un futuro,
mediante técnicas de mejoramiento genético.

6
CAPÍTULO I
ANTECEDENTES
Durante los últimos años se ha observado un gran interés en la producción de
energía derivada de fuentes renovables, debido al notorio agotamiento de los
combustibles fósiles y al impacto que generan en el medio ambiente. Entre estas fuentes
renovables se encuentran los biocombustibles. El término biocombustible se refiere a
cualquier combustible, ya sea en estado sólido, líquido o gaseoso, que se obtenga a partir
de la desintegración de materias primas biológicas o vegetales, se les conoce también
con el nombre de combustibles renovables, ya que son producidos a partir de materias
primas con estas características.
La producción de biocombustibles tiene el potencial de reducir la dependencia de
los combustibles fósiles, contribuyendo así a la seguridad energética de los países
importadores de petróleo. Además, los biocombustibles pueden beneficiar al sector
agrícola, incluyendo al sector de pequeña producción, a través de la generación de
empleo e ingresos. Entre los cultivos energéticos hay grandes diferencias en cuanto a su
potencial.
Por medio de la fotosíntesis, la radiación solar se transforma en esqueletos
carbonados, que pueden ser utilizados como combustible, directamente o después de ser
procesados. En sus formas tradicionales, el uso de la bioenergía se pierde en los
orígenes de la humanidad. Sin embargo, en los últimos años, los biocombustibles líquidos
están siendo considerados como una fuente energética renovable, capaz de atenuar los
problemas ambientales producidos por el uso de los combustibles fósiles.
J. curcas es una planta originaria de América Central y cultivada en todo el mundo,
cuyas semillas contienen un aceite que se puede convertir en biodiésel. Esta planta,
resiste en un alto grado la sequía y prospera con apenas 250 a 600 mm de lluvia al año.
Además, puede crecer en suelos marginales y no precisa de un uso importante de
pesticidas ni fungicidas, tiene un elevado rendimiento por hectárea y no es comestible por
lo que su utilización para la fabricación de biodiésel no contribuye al aumento de los
precios de los cultivos destinados a la alimentación (como si pasa por ejemplo con el
maíz, la soja, etc.).

7
1. Fuentes y producción de aceite
Los aceites derivados de vegetales y de algas constituyen la mayor parte de las
potenciales fuentes para la producción de biodiésel. En la tabla 1.1 se muestra la
composición en el contenido de ácidos grasos de aceites de fuentes prometedoras, tanto
de plantas como animales, para la producción de biodiésel.
La mayoría de los aceites vegetales tienen un perfil prometedor de ésteres de
ácidos grasos y valores de cetano de alrededor de 60 (véase Tab. 1.2). El aceite vegetal
más frecuente para la producción de biodiésel, en todo el mundo, es el de colza,
comúnmente llamado canola.
Un estudio del desempeño de la producción de biodiésel de J. curcas en China,
indica que aunque los costos asociados a las etapas de transesterificación y distribución
son bajos, el sistema no resulta económicamente factible debido a que los costos de
recolección y tratamiento de las semillas suman el 83% de los costos totales, tal y como
se muestra en la figura 1.1 [4], (la capacidad de producción contemplada en este estudio
es de 25,800 m3 al año). En un estudio paralelo realizado en la India se estimaron que los
costos de producción de las semillas de J. curcas representan de un 83% a un 84% de los
costos totales y, en consecuencia, el precio de venta del biodiésel resultaba un 11.11%
más elevado al del petrodiésel [5].

8
Tabla 1.1. Contenido de aceite y composición de ácidos grasos de varias oleaginosas [5].
Tabla 1.2. Producción de aceite de origen vegetal [5].
Fuente de aceite Producción
(gal/acre/año) Área terrestre
(M) % de U. S. como
superficie de cultivo
Microalgas 6280 10 2
Árbol de china 700 85 20
Palma aceitera 635 94 22
Cocotero 287 209 50
Jatropha 202 297 71
Castor aceitero 151 397 95
Aceite de olivo 129 465 111
Aceite de colza 127 472 112
Aceite de semilla de amapola
124 484 115
Aceite de cacahuete 113 531 126
Aceite de girasol 102 588 140
Fuente Contenido de aceite
Valor de yodo
(aceite)
Índice de cetano
(ésteres)
Palmítico (16:00)
Palmitoleico (16:1)
Estérico (18:00)
Oleico (18:1)
Linoleico (18:2)
Linolénico (18:3)
Canola 30 98 55 3.5 - 0.9 64.4 22.3 8.2
Olivo 20 81 60 9.2 0.8 3.4 80.4 4.5 0.6
Aceite de girasol
47 125 52 6.0 - 4.2 18.7 69.3 -
Soya 18 130 53 10.6 - 4.8 22.5 52.3 8.2
Palma aceitera
35 54 65 47.9 - 4.2 37.0 9.1 0.3
Maíz - 120 53 11.8 - 2.0 24.8 61.3 -
Ricino - 85 - 1.1 - 3.1 4.9 1.3 -
Coco 35 10 70 9.7 0.1 3.0
6.9 2.2 -
Jatropha curcas
- 185 - 13.3 1.0 4.9 32.0 45.0 0.2
Camelina 155 - 5.4 - 2.6 14.3 14.3 38.4
Almendro 54 - - 6.5 0.5 1.4 70.7 20.0 -
Algodón 40 105 65 47.9 - 4.2 37.0 9.1 0.3

9
Aceite de árbol de tung
100 600 143
Aceite de salvado de arroz
88 682 162
Aceite de cártamo 83 723 172
Aceite de semillas sésamo
74 811 193
Aceite de linaza 51 1176 280
Aceite de avellana 51 1176 280
Aceite de soja 48 1250 298
Aceite de cáñamo 39 1538 366
Aceite de algodón 35 1714 408
Kenaf 29 2069 493
Aceite de maíz 18 3333 794
Figura 1.1. Distribución de los costos de producción de biodiésel a partir de J. curcas [6].

10
1.1. Jatropha curcas
J. curcas pertenece al grupo de plantas de las oleaginosas, un conjunto de plantas
que contiene más de 3,500 especies agrupadas en 210 géneros. Se le encuentra
mayormente a bajas elevaciones, por debajo de los 1,200 metros sobre el nivel del mar
(msnm), tanto en áreas secas, como húmedas, en planicies o colinas, con precipitaciones
de 300 a 1,800 mm por año y temperaturas de 18 a 28 °C, si bien se le puede sembrar en
sitios con temperaturas de hasta 34 °C [3].
Figura 1.2. Mapa geográfico de Jatropha en México.
El piñón, nombre común con el que es conocida en México J. curcas, además, es
una planta perenne cuyo ciclo productivo se extiende de 45 a 50 años, posee un
crecimiento rápido con una altura normal de 2 a 3 m, presenta una raíz principal y dos
periféricas o secundarias. El tallo es erguido con un diámetro máximo de 20 cm, posee
ramas gruesas que crecen de forma discontinua en cada incremento y en las hojas se
forman de 5 a 7 lóbulos acuminados, poco profundos y con pecíolos largos de alrededor
de 10 a 15 cm. Las inflorescencias se forman en la parte terminal de las ramas, las flores,
tanto femeninas como masculinas, son pequeñas, de 6 a 8 mm de diámetro y presentan
microvellosidades (pubescentes).
En México, la planta se encuentra en forma silvestre en diversos estados de la
República Mexicana, como Tamaulipas, Veracruz, Tabasco, Yucatán, Chiapas, Oaxaca,

11
Quintana Roo, Guerrero, Michoacán, Sinaloa, Sonora, Puebla, Hidalgo y Morelos, pero
sólo es utilizada de manera tradicional por los pobladores de la región de Papantla,
Veracruz y Puebla (véase Fig. 1.2).
De las semillas de J. curcas se extrae el aceite susceptible de ser procesado y
transformado en biodiésel. Del tallo se extrae látex, y de sus hojas y cortezas otras
sustancias para aplicaciones medicinales.
1.1.1. Clasificación taxonómica
El género Jatropha comprende aproximadamente 175 especies de hábitos
suculentos, arbustos y árboles (algunos son caducifolios, como J. curcas). Según
Cronquist [7] la clasificación taxonómica de J. curcas es:
Clasificación taxonómica
Reino Plantae
Filo/división Magnoliophyta Clase Magnoliopsida Orden Euphorbiales Familia Euphorbiaceae Especie curcas
Jatropha tiene diferentes nombres comunes, dependiendo del lugar: Coquito, Capate,
Tempate, Piñón, Piñoncito, Piñol, Higos del Duende, Barbasco, Piñones Purgativos, y
Periyanasi (piro) [8].
1.1.2. Morfología vegetal
J. curcas es un arbusto o árbol pequeño, de 2 a 6 m de altura, con corteza blanco-
grisácea, que exuda un látex translúcido (véase Fig. 1.3).
Raíz. Normalmente se forman cinco raíces de los arbolillos, una central y cuatro
periféricas.

12
Hoja. Las hojas de 9 a 15 cm de ancho se forman normalmente con 5 a 7 lóbulos
acuminados, poco profundos y grandes; tienen pecíolos largos con una longitud de 10 a
15 cm.
Tallo. Los tallos crecen con una discontinuidad morfológica en cada incremento, es un
cilindro verde robusto que produce ramas con savia láctea o rojiza viscosa [9].
Figura 1.3. Partes importantes de la planta. a) Floración en la rama, b) corteza, c) limbo de la hoja,
d) pistilo de la flor, e) flores, f) corte transversal de la fruta inmadura, g) frutas, h) corte longitudinal
de los frutos, i) semilla [10].
Flor. Las inflorescencias se forman en el lado axial de las hojas, en las ramas, formando
una inflorescencia compuesta a manera de panícula, en la cual el eje principal se ramifica
una o más veces. Ambas flores, masculinas y femeninas, son pequeñas (6-8 mm),
verdoso-amarillo en el diámetro y pubescente. Los pétalos son de 6-7 mm de largo. La
longitud del pecíolo varía entre 6- 23 mm [11].
Fruto. Son cápsulas drupáceas y ovoides, después de la polinización, se forma una fruta
trilocular de forma elipsoidal. Las frutas son cápsulas inicialmente verdes que cambian a
café oscuro o negro conforme maduran. Las cápsulas de los frutos son de 2.5 a 4 cm de
largo por 2 cm de ancho, elipsoidales y lisas que cuando maduran cambian a amarillas.

13
Semilla. La fruta produce tres almendras negras, cada una de aproximadamente 2 cm de
largo y 1 cm de diámetro. La semilla se cosecha cuando la cápsula está madura, lo cual
ocurre después de dos a cuatro meses de la fertilización [11].
1.1.3. Importancia del cultivo
Las plantas que acumulan aceite tienen mejores posibilidades de emplearse como
fuente de material para producir Bioenergéticos que las que producen sacarosa o
almidón, ya que su transformación es más simple. De las diversas plantas que acumulan
aceite, el piñón muestra características que la colocan como una de las principales
alternativas para emplearse en la producción de biodiésel.
Las semillas contienen alrededor de 38% de aceite en peso, aunque hay autores
que reportan cifras de hasta aproximadamente 50% [12] (Figs. 1.4 y 1.5). De esta cifra se
puede obtener entre 27% y 32% del aceite usando máquinas extractoras a presión en frío
[12]. Este aceite es usado principalmente para la producción de jabones, insecticidas y
como combustible en forma de aceite puro o después de ser transesterificado, como
biodiésel para ser usado en motores, cocinas y faroles para el alumbrado público. Como
biodiésel se emplea en mezclas B2, B5, B10, B20 o puro.
Figura 1.4 A. Semillas. B. Frutos inmaduros de J. curcas.

14
Las principales cualidades de la planta son:
Puede crecer en casi cualquier tipo de tierra.
Puede sobrevivir períodos largos de sequía, por lo que puede cultivarse en áreas
con bajo régimen de lluvia (250 mm por año).
Su propagación es fácil de implementar.
Produce frutos desde el primer año, su producción se estabiliza al quinto año y
continúa durante 25 - 30 años, produciendo frutos de buena calidad.
Produce diversos productos y subproductos que pueden ser aprovechables.
El contenido de aceite se encuentra dentro del orden del producido por
otras oleaginosas.
Este aceite no tiene aplicaciones para la industria alimentaria, por lo cual su único
uso es el industrial. (Figura 1.4 y 1.5).
Figura 1.5. A) Inflorescencia de J. curcas que contiene tanto flores masculinas como femeninas; B.
Arbusto; C. Semillas maduras y fruto abierto; D) Semillas maduras de J. curcas; E. Hojas foliares.

15
1.1.4. Componentes químicos
El género Jatropha contiene, entre otros compuestos químicos: alcaloides,
sapogeninas, taninos, ésteres, toxoalbúminas, y compuestos cianogénicos. De particular
importancia son los ésteres de forbol que se encuentran presentes en las semillas, lo que
le da parte de la toxicidad a la semilla (véase Tab. 1.3).
Tabla 1.3. Composición química del aceite de Jatropha.
Ácidos grasos
Teórico (%) [3]
Mirístico 0 - 0.1 Palmítico 14.1 - 15.3
Esteárico 3.7 - 9.8
Palmitoleico 0 - 1.3
Oleico 24.3 - 45.8
Linoleico 29.0 - 44.2
Ricinoleico 0 - 0.3
Araquídico 0 - 0.3
Behénico 0 - 0.2
1.1.5. Ambiente o localidad
Latitud: 28º N a 30º S.
Altitud: las procedencias han sido reunidas de altitudes que varían de 7 a 1,600 msnm.
Temperatura: las temperaturas medias de los sitios de colección de procedencia van de
20 a 36 ºC. Puede sobrevivir una corta y ligera escarcha pero se encuentra mejor a
temperaturas elevadas.
Clima: tropical, cálido-húmedo, y templado.
Agua: la precipitación media anual de sitios de colección es de 520 a 2,000 mm. Sin
embargo, la especie sobrevive bien en las regiones semiáridas.
Radiación: rango e intensidad: la luz del sol luminosa. Fotoperiodo: es insensible a la luz
del día.

16
Suelo: crece en las tierras bien drenadas, con buena aireación y se adapta bien a las
tierras marginales con un volumen de nutrientes bajo. Franco-arenoso o arcillo-arenoso
con abundante materia orgánica, profundo y bien drenado porque es susceptible a
inundaciones.
J. curcas requiere de una precipitación media anual de entre 600 mm a 1,200 mm;
además, por su naturaleza tóxica, los cultivos de esta planta destinados a la producción
de biodiésel no resultan de la industria que compite con la agricultura. México se
considera como una región geográfica muy significativa para esta planta ya que se
encuentra en el cinturón norte de los suelos y en condiciones climáticas aptas para su
plantación, como se puede apreciar en la figura 1.6.
Figura 1.6. Condiciones climáticas más favorables para el cultivo de J. curcas y de la palma de
aceite.
1.2. Biosíntesis de lípidos en plantas
En las plantas, la biosíntesis de los ácidos grasos ocurre exclusivamente en los
plástidos y es iniciada por la enzima acetil CoA carboxilasa [13], la cual convierte al acetil
CoA a malonil CoA. Posteriormente, el grupo malonil es transferido a una proteína
transportadora de acilos (ACP) formando al malonil-ACP. Este último entra a una serie de
reacciones de condensación, deshidratación y reducción que resulta en la adición de dos
moléculas de carbono a la cadena de acilo por cada ciclo, culminando con la formación de
los ácidos grasos saturados palmitato (16:0) y estearato (18:0). Por último, los ácidos
grasos son desaturados por la adición de uno o más doble enlaces, el primer doble enlace

17
es introducido por la enzima estearoil ACP desaturasa (SAD) [14] (como se muestra en la
Fig. 1.7).
La biosíntesis de lípidos es afectada por factores ambientales, los cuales alteran
sus niveles en las plantas, éstos comprenden la luz, la temperatura, el estrés hídrico, los
constituyentes del suelo, factores atmosféricos y factores como el daño físico y el ataque
de patógenos [15].
La acetil-CoA es un precursor para diversas moléculas y se encuentra en múltiples
organelos. El primer paso comprometido de la biosíntesis de los ácidos grasos es la
conversión de acetil-CoA en malonil-CoA por la acetil-CoA carboxilasa y luego a malonil-
ACP (proteína transportadora de acilos) por una transacilasa. En la mayoría de los tejidos,
de los organismos eucariontes, incluyendo a las plantas, el malonil-ACP se alarga durante
ocho ciclos, dos unidades de carbono a la vez, a través del complejo de la ácido graso
sintasa, hasta palmitoil (16:0) -ACP (o-CoA). El complejo ácido graso sintasa implica
cuatro reacciones enzimáticas diferentes con cada ciclo, iniciando con una condensación,
seguida por una reducción, una deshidratación y una segunda reducción.
Las reacciones de condensación son catalizadas por las enzimas 3-cetoacil-ACP
sintasas o KASs. La primera reacción de condensación que va desde acetil-CoA a 3-
cetobutirato es catalizada por la KAS III, la reacción butiril-ACP (C4) a palmitoil-ACP (C16)
por KAS I, y a palmitoil-ACP estearoil-ACP (Cl8) por KAS II. La reacción se detiene en
ácidos grasos C16 y C18, no sólo en virtud de la especificidad de las enzimas KAS, sino
también por la acción de las tioesterasas (TES), éstas son enzimas que hidrolizan los
enlaces acil-S-ACP tioéster. El aceite se sintetiza durante la segunda etapa de
maduración de la semilla [15] momento en el cual los genes que codifican para las
enzimas biosintéticas son altamente expresados.

18
Figura 1.7. Síntesis de los principales ácidos grasos saturados, insaturados y acil-CoA en las
células vegetales. Los grupos acilo de ácido graso son nombrados por el número de carbonos:
número de dobles enlaces 2:00: acetilo; 3:00: malonil; 4:0: butiril; 16:00: palmitoil (hexadecanoato);
18:0: esteárico (octadecanoato); 18:1: oleoil (octadecenoato); ACP: proteína acil portador; ACS:
acil-CoA-sintetasa; KAS: 3-cetoacil-ACP sintasa, TE: tioesterasa.
La biosíntesis de los ácidos grasos en las plantas se lleva a cabo en los plástidios,
no en el citosol, por lo que se requiere de un mecanismo para moverlos desde los
plástidios. En tanto que en levaduras y animales, la acetil-CoA se produce en la matriz
mitocondrial, mientras que el resto de la biosíntesis de los ácidos grasos es un proceso
citosólico.
1.2.1. Enzimas claves en la biosíntesis de los ácidos grasos
La enzima acetil-CoA carboxilasa, utiliza acetil-CoA para generar malonil–CoA
[13;15]. Existen dos diferentes isoformas de esta enzima: (i) la tipo I u homomérica, la cual
consiste de un solo péptido que contiene tres dominios funcionales: el de la proteína
transportadora de carboxil biotina (BCCP), el dominio carboxil transferasa (CT) y la biotina
carboxilasa [16;17] y la tipo II o heteromérica, que consta de cuatro polipéptidos
separados: BCCP, BC, CTα y CTβ [18]. El péptido CTβ es codificado por el genoma del
cloroplasto y los otros tres péptidos son codificados en el genoma nuclear [19].
El malonil-CoA generado por la forma heteromérica es esencial para la síntesis de
los ácidos grasos en los plástidos [20]. En cambio, el malonil-CoA generado por la forma
homomérica generalmente es encontrado en el citosol y es requerido para la síntesis de

19
flavonoides y ácidos grasos de cadena larga. Esta enzima, que es clave para la
biosíntesis de lípidos, es altamente regulada a nivel transcripcional, post transcripcional y
de actividad enzimática [19] (como se muestra en la Fig. 1.8).
Figura 1.8. Precursores de la síntesis de los ácidos grasos.
Tomando en cuenta la importancia de la biosíntesis de los ácidos grasos, otra
enzima, que además presenta una estrecha relación con la aclimatación a bajas
temperaturas de las plantas, es la estearoil-ACP desaturasa (SAD), la cual se localiza en
el estroma de los plástidos y se encarga de catalizar la desaturación del estearoil-ACP a
oleoil-ACP. SAD cumple una función muy importante en determinar la proporción de
ácidos grasos saturados/insaturados en las plantas y esta proporción está relacionada
con diversas funciones, especialmente con la aclimatación de las plantas a bajas
temperaturas [21]. Por ejemplo, en estudios realizados en papa (Solanum tuberosum L.),
se ha determinado que el incremento en las insaturaciones de los ácidos grasos de la
membrana, que aumenta la tolerancia al frío, está relacionado con un aumento en la
expresión del gen SAD [22].
Costa et al. [23] determinaron un gran número de ESTs que codifican para casi
todas las enzimas de la ruta de biosíntesis de los ácidos grasos (excepto KAS III y HAD)
(Figura 1.9), es decir, enzimas que catalizan reacciones que producen los ácidos
palmítico, linoleico, oleico y esteárico (FATA, PCH y FatB, tabla 1.4) los principales
componentes del aceite de las semillas de Jatropha. Además, determinaron 12 ESTs de
la oleoil-ACP desaturasa (FAD2, Tabla 1.4), que cataliza la poliinsaturación de la oleoil-

20
ACP (18:01) a linoleoil-ACP (18:02). Considerando que el ácido oleico y linoleico son los
principales constituyentes del aceite de Jatropha, esta enzima es un importante objetivo
biotecnológico para la modulación de la composición de los ácidos grasos presentes en
los triacilglicéridos que conforman el aceite de Jatropha.
Tabla 1.4. Enzimas involucradas en la biosíntesis de los ácidos grasos que forman el
aceite en las semillas de J. curcas.
Enzimas relacionadas con la acumulación de aceite en semillas
Símbolo Enzima Unisecuencias
FatA Acil-ACP tioesterasa A 2
FatB Acil-ACP tioesterasa B 1
ACC Acetil-CoA carboxilasa 4
EAR Enoil-ACP reductasa 2
HAD Hidroxiacil-ACP dehidrasa 0
KAR Cetoacil-ACP reductasa 5
KAS I Cetoacil-ACP sintasa I 4
KAS II Cetoacil-ACP sintasa II 2
KAS III Cetoacil-ACP sintasa III 0
MAT Malonil-CoA ACP transaciclasa 1
FAD2 Oleoil-ACP desaturasa 2
PCH Palmitoil-CoA hidrolasa 1
SAD Estearoil-ACP desaturasa 1

21
Otra clase importante de lípidos son los acilgliceroles, que actúan como una
reserva de energía en muchos organismos y son los principales componentes del
almacenamiento de aceites en semillas. El acilglicerol más común en los aceites de
semillas es triacilglicerol (TAG).
Figura 1.9. Ruta de biosíntesis de los ácidos grasos en Jatropha. Los íconos al lado de cada
nombre de la enzima muestran el número total de ESTs correspondientes a esa enzima en JD y
JG, por separado. KAS I participa desde el 2º al 6º ciclo de extensión KAS II participa en el 7º ciclo
de extensión de la cadena de carbonos. Ambos FatA y FatB pueden catalizar la conversión de
18:0-ACP en ácido esteárico. Los ácidos grasos producidos por esta vía son 16:0 (ácido palmítico),
18:0 (ácido esteárico), 18:1 (ácido oleico) y 18:2 (ácido linoleico) [23].
1.2.2. Ácido graso sintasa.
Los ácidos grasos insaturados en las plantas son relacionados con las funciones
de muchas de las plantas, especialmente en la aclimatación a baja temperatura [21]. La
desaturación de ácidos grasos de la planta fuera del plástido (en el citoplasma) parece
estar limitada a oleato, que se puede desaturar a linoleato (18:2) y linolenato (18:3).
Además, dependiendo de la planta, el oleato se puede modificar adicionalmente por
alargamiento (hasta 20:1, 22:1, y/o 24:1), o por adición de grupos funcionales. Estos

22
ácidos grasos, junto con los ácidos grasos saturados palmitato y estearato, se pueden
entonces ensamblar en triglicéridos.
En las bacterias y otros procariotes, las enzimas involucradas en la biosíntesis de
ácidos grasos, se encuentran separadas y funcionan únicamente como un complejo
enzimático en donde cada producto es cedido a la enzima siguiente para proseguir con su
metabolismo.
El Butirill-ACP puede después condensarse con otra molécula de malonil-ACP,
después de siete vueltas de este ciclo se produce palmitoil ACP, el cual es siempre el
producto final en este mecanismo de síntesis (véase Fig. 1.10). Ningún intermediario es
liberado por el complejo enzimático antes de formar palmitoil-ACP (véase Tab. 1.5). La
hidrólisis del Palmitoil-ACP dará ácido palmítico que es enseguida convertido a Palmitoil-
CoA [20].
Figura 1.10. Ruta de síntesis del complejo enzimático ácido graso sintasa.

23
Tabla 1.5. Complejo Proteico de la ácido graso sintasa.
El alargamiento final de 16:00-ACP a 18:00-ACP se lleva a cabo en el plastidio por
KASII [20]. Los compuestos principales de alargamiento, ácidos grasos C16 y C18, puede
ser aún más desaturados en la membrana plastídica y el retículo endoplásmico para
producir ácidos grasos insaturados [24] (Fig. 1.12).
Los genes de KASII se han clonado y caracterizado en varias especies vegetales,
como Arabidopsis thaliana [25], Arachishy pogaea [24], Perilla frutescens, Betula pendula,
Cuphea lanceolata, Cuphea wrightii y Jessenia bataua.
En el caso de J. curcas, un árbol multipropósito arbusto/pequeño perteneciente a
la familia de las Euphorbiaceae [26], que puede crecer bien bajo condiciones climáticas
adversas, debido a su bajo contenido de humedad, fertilidad, y tolerancia a las altas
temperaturas, ha recibido mucha atención por su alto contenido de aceite en sus semillas
y su alto rendimiento de aceite por unidad de superficie, que sólo es superado por el
aceite de palma [26;27].
En esta especie se han modificado algunos genes como KASII, dando como
resultado una alteración en la composición de ácidos grasos [28]. En el 2012 Wei y
colaboradores aislaron el ADNc (1722 pb) que codifica para una proteína portadora β-
Cetoacil-acil (ACP) sintasa II (KASII) una de las principales enzimas involucradas en la
biosíntesis de ácidos grasos. Una vez aislado el ADNc de KASII de Jatropha realizó una
sobreexpresión en la planta Arabidopsis thaliana, utilizando el promotor 35S del virus del

24
mosaico de la coliflor, obteniendo una disminución en el contenido ácido graso de 16-C y
un aumento considerable en la producción de 18-C, expresándose tanto en hoja como en
semillas de Jatropha [29].
1.2.3. Producción de biodiésel de J. curcas
Existen diversas plantas que podrían ser potenciales materias primas para la
producción de biodiesel. El rendimiento de cada una de ellas, es decir, la cantidad de
biodiesel que se puede extraer por cada hectárea de plantación por año, se encuentra en
la tabla 1.6.
Tabla 1.6. Rendimiento de los posibles insumos para la producción de biodiesel.
Insumo Rendimiento
(
)
Soja 420
Arroz 770
Tung 880
Girasol 890
Maní 990
Colza 1100
Ricino/Tartago 1320
Jatropha curcas
1590
Aguacate 2460
Cocotero 4200
Palma aceitera
5550
Fuente: http://www.engormix.com/MA-agricultura/cultivos-tropicales/articulos/biodiesel-energia-vegetal-petroleo-
t862/078-p0.htm,

25
Aunque la J .curcas tiene un rendimiento menor que el de la palma aceitera, el
cocotero o el aguacate, esta planta tiene ciertas ventajas que incrementan el interés en su
uso como materia prima para la producción de biodiesel. La diferencia radica que la J.
curcas es un arbusto y, por tanto, su cultivo es más fácil que el de los árboles y su periodo
de gestación, más corto; la planta requiere de pocas cantidades de insumos (agua,
fertilizantes y de pesticidas) en comparación con plantas comestibles, es resistente a
pestes comunes y provee subproductos que pueden ser aprovechables como abonos y
madera.
Debido al elevado contenido de ácidos grasos libres presente en el aceite crudo de
J. curcas (un 14% recién extraído y puede aumentar a un 21% cuando es almacenado por
un largo periodo de tiempo) éste debe ser sometido a un proceso de esterificación previo
a la transesterificación [30;31]. Para ello se han realizado estudios en torno a las mejores
condiciones de esterificación del aceite de J. curcas (Tab.1.7).
Tabla 1.7. Condiciones óptimas para la esterificación del aceite de J. curcas.
Tasa metanol / aceite [32]
Concentración del catalizador
Temperatura (°C)
Tiempo Fuente
3:7 1% H2SO4 (p/p) 65±0.5 180 [33]
6:1 0.5% H2SO4 [32] 40 120 [34]
60% p/p 1% H2SO4 (p/p) 50 60 [35]
28% v/v 1.43% H2SO4 [32] 60 88 [36]
60% p/p 1% H2SO4 (p/p) 60 180 [37]
*Estos tratamientos son para aceites que han sido almacenados prolongadamente y tienen más de
un 20% de ácidos grasos libres.
Las condiciones óptimas para la transesterificación del aceite de J. curcas
esterificado también han sido abordadas por diversos estudios. Los principales
catalizadores considerados son hidróxido de sodio e hidróxido de potasio.
El aceite crudo de J. curcas ha sido sometido directamente al proceso de
transesterificación empleando el n-hexano extractor como co-solvente, lo que dio como

26
resultado un biodiésel que cumplía con las normas de calidad correspondientes; sin
embargo, este proceso es aun de carácter experimental [38].
El aceite de Jatropha, contiene predominantemente ácidos linoleico y oleico, los
cuales constituyen del 72 al 80% de los ácidos grasos totales [39] [40]. Otros ácidos
grasos presentes en el aceite incluyen los ácidos palmítico y estéarico [41], así como los
ácidos caprílico, mirístico, palmitoleico, araquídico, behenico y lignocérico [42].
Es de especial interés el hecho de que el contenido de los ácidos grasos
saturados, palmítico y esteárico varía entre 17.8 y 25.1%, en tanto que el contenido de los
ácidos grasos insaturados, palmitoleico, oleico y linoleico varía entre 51.8 y 96.7% [43].
La biotecnología puede ayudar a acelerar la selección de variedades que sean
más apropiadas para la producción de biocombustibles con un aumento en la producción
de biomasa por hectárea, aumento en el contenido de aceites o de azúcares
fermentables, o que mejoren las características de procesamiento que faciliten su
conversión a biocombustibles-.
Los campos de la genómica, proteómica y metabolómica jugarán una creciente e
importante función en ello. La llegada de la ingeniería genética trajo consigo nuevas
estrategias para el mejoramiento genético de las plantas, permitiendo la introducción
directa de genes que codifican para el atributo de interés [44]. Este hecho no solo permite
una incorporación más precisa de la información genética en la planta de interés
(comparado con la transferencia variable y sin control que se lleva a cabo durante la
recombinación sexual), pero también evade la necesidad de compatibilidad sexual entre
las líneas parentales [45]. Así es posible modificar genéticamente a una planta con genes
provenientes de cualquier organismo, no sólo con los genes relacionados a la especie
[44].
Así, el potencial de introducir rasgos novedosos en cultivos de plantas superiores,
ha permitido considerar el concepto de semilla de aceite diseñada, a la cual se le han
añadido características no presentes en los cultivos actuales (tales como soya, maíz,
brásica, etc.) [46]. Tales semillas podrían contener ácidos grasos que sean útiles en
procesos industriales, ya sea como lubricantes o como materia prima para otros usos [47].

27
La transformación mediada por Agrobacterium tumefaciens, se ha convertido en el
método más usado en la investigación de plantas, y por ende en una de las principales
tecnologías para la generación de plantas transgénicas [48]. Para aplicar este método a J.
curcas, primero se debe establecer un sistema eficiente de regeneración.
1.2.4. Necesidad de transformar J. curcas
Existen diversas razones para realizar este tipo de estudios y una de las
principales es la adopción de la ingeniería genética en este tipo de cultivo. La adopción
del sistema genético en J. curcas, tiene la finalidad de obtener varios métodos de
transformación genética y en paralelo aprovechar los sistemas de transformación para
obtener mayor cantidad de frutos, obtener plantas resistentes a la salinidad y bajas
temperaturas, mejorar las propiedades medicinales, bajar la toxicidad de algunas
especies de J. curcas, crear resistencia al estrés biótico y elevar la eficiencia de
germinación. La razón más fuerte, radica en modificar el contenido de los ácidos grasos
de las semillas y con ello modificar la cantidad de aceite que se obtiene de las semillas o
para mejorar la calidad del mismo y estas modificaciones permitirán obtener biodiesel a
partir de J. curcas y no depender demasiado de los yacimientos fósiles.
Debido al largo periodo de germinación de J. curcas y al hecho de que este cultivo
todavía no es importante en la alimentación humana, algunos científicos dieron inicio a los
estudios de modificación genética de esta planta, contemplando inicialmente los sistemas
de regeneración que van a permitir la incorporación de los sistemas de transformación
genética [24;49].
Recientemente, se han publicado varios protocolos para la transformación de J.
curcas [50]; [51]. Sin embargo, no en todos se han regenerado plantas transgénicas.
La transformación de J. curcas, se ha llevado a cabo con A. tumefaciens [51] y
mediante el bombardeo con partículas recubiertas con el plásmido pBI426 conteniendo
una fusión de GUS-ntpII bajo el control de un promotor doble 35S. Se han transformado
diferentes tejidos como hoja meristemos apicales [51] y cotiledones.

28
1.2.5. Transformación genética en J. curcas
El mejor sistema de transformación genética se basa en obtener el plásmido que
tenga alta frecuencia de inserción en el genoma de J. curcas. El plásmido preferido por
los grupos de investigación es el pCAMBIA1301 y en paralelo se ha realizado el estudio
de los plásmidos pCAMBIA2301, pBI121, pBI426 y pIG121Hm. Estos últimos no han
cubierto la mayoría de las expectativas de los trabajos de transformación genética y por lo
tanto no se emplean mucho [52]. Otra de las herramientas moleculares que son
esenciales en la transformación mediada por A. tumefaciens es la cepa bacteriana; la
cepa de A. tumefaciens que ha sido preferida por los grupos científicos es la LBA4404 y
en paralelo se ha realizado la comparación con las cepas EHA105 y EHA101; estas
últimas no han sido las preferidas en los trabajos de transformación genética de J. curcas.
Por otro lado, una vez realizada la infección por A. tumefaciens es importante emplear
marcadores de selección para obtener las posibles transformantes.
En la gran mayoría de los trabajos se emplea kanamicina como primera opción en
la selección de las transformantes, en otros casos se usa la higromicina. Uno de los
principales problemas que enfrentan los sistemas in vitro de regeneración de J. curcas en
la transformación genética, es eliminar A. tumefaciens en el cultivo in vitro [24;53].
En la tabla 1.8, se presenta un resumen de las características más importantes
durante el proceso de transformación de J. curcas, los cuales han sido utilizados para
obtener y reducir el contenido de ácidos grasos.
El conocimiento total de los genes involucrados en la ruta biosíntetica de los
ácidos grasos podría combinarse con la biología sintética y aprovechar algunos
microorganismos facilitadores de la biotecnología para producir masivamente bioenergía.
Las repercusiones de estos aspectos moleculares, se verá reflejado en una mayor
cantidad de semillas de J. curcas, aumento en la producción del aceite y una buena
calidad del biodiésel [54;55].

29
Tabla 1.8. Resumen general de trabajos reportados en transformación de J. curcas.
JC. Cotiledones jóvenes; TGI. Integración del gen diana; PR. Regeneración de planta; RNAi, ARN de
interferencia; AS, anti-sentido; CL, hoja cotiledón; LE. Explantes de hoja; E. embrión; TE. Expresión génica
transitoria; SR. regeneración de brotes; LS. Segmentos de hoja; SA. Segmentos de hipocótilo; YL. Hojas
jóvenes; SA. Ápices

30
HIPÓTESIS
Si bien es cierto que Jatropha puede sobrevivir en condiciones poco favorables para la
agricultura, sus rendimientos en producción de aceites son muy bajos. Entonces, para que
J. curcas se considerada como el cultivo energético del futuro, será necesario analizar las
los principales genes involucrados en la ruta de biosíntesis de ácidos grasos de las
semillas de J. curcas para que produzcan aceites que permitan obtener biodiesel de mejor
calidad.
OBJETIVO GENERAL
Analizar la expresión del gen JcKASII en frutos maduros e inmaduros y desarrollar un
protocolo de transformación vía A. tumefaciens en cultivos in vitro de J. curcas.
OBJETIVOS ESPECÍFICOS
I. Analizar las secuencias de KASII en otras especies y la expresión basal de KASII en
los frutos de J. curcas.
II. Obtener una línea de callos embriogénicos para J. curcas.
III. Establecer un protocolo de transformación vía A. tumefaciens en cultivos in vitro de
J. curcas.
IV. Determinar el efecto del DMSO y DMFM en la tinción de GUS en los tejidos
transformados.
JUSTIFICACIÓN
La introducción precisa y controlada de genes específicos deseables, así como la
inactivación de genes específicos indeseables, permite la posibilidad de modificar la
composición de los ácidos grasos presentes en el aceite de la planta. De este modo, se

31
podrían introducir en la planta de J. curcas nuevos rasgos completamente diferentes, que
hagan más viable el uso de su aceite para la producción de biodiésel. La sobreexpresión
de KASII en J. curcas, es posible que modifique la composición de los ácidos grasos
presentes en la semilla, generando un aumentando en los niveles de los ácidos grasos
saturados. Este aumento tiene el potencial de mejorar la calidad del biodiésel obtenido del
aceite de Jatropha.

32

33
CAPÍTULO II
MATERIALES Y MÉTODOS
Estrategia experimental
Para cumplir con los objetivos propuestos, se diseñó la estrategia experimental
ilustrada en la figura 2.1. Se decidió dividir el proyecto en cuatro fases de investigación:
Fase 1. Para cumplir con el objetivo 1; se realizó la búsqueda de secuencias in
silico KASII de J. curcas.
Fase 2. En esta fase se complementará el objetivo 1. Durante esta etapa se
procedió a efectuar los ensayos in vivo, colectando material vegetal (hojas y semillas) de
la plantación experimental, para determinar la expresión basal del gen KASII en J. curcas.
Fase 3. En esta etapa, se cumplirá con el objetivo 2, donde se establecieron las
condiciones optimas para obtener callos a partir de explantes de J. curcas.
Fase 4. Aquí se cumplirán los objetivos 3 y 4. En el cual se estableció el protocolo
de transformación de callos de J. curcas vía Agrobacterium tumefaciens con los
plásmidos pCAMBIA2301 el cual contiene el gen reportero uidA. Además, de ver el efecto
del DMSO y DMFM en los explantes transformados durante la prueba de localización
histoquímica de GUS.

34
Figura 2.1. Estrategia experimental.

35
2.1. INTRODUCCIÓN
Las células eucariontes responden al crecimiento, desarrollo y factores
ambientales, en parte por la regulación de la expresión de grupos específicos de genes.
Según Reddy y Subba [56], las plantas están expuestas constantemente a factores
estresantes que limitan su crecimiento y desarrollo, por ejemplo, temperatura baja o alta,
sequia, alta salinidad (factores abióticos) o patógenos como bacterias, hongos o virus
(factores biótico).
La regulación de la biosíntesis de proteínas, es una de las principales formas que
poseen los organismos para controlar su metabolismo. La respuesta de un organismo a
nivel molecular puede depender de factores ambientales que ejerzan su acción sobre las
células del mismo, cada célula debe ser capaz de modular una respuesta encendiendo o
apagando genes de forma coordinada.
La importancia de elegir a KASII radica en el hecho de ser la responsable de
condensar al palmitoil-ACP con malonil-ACP para formar estearoil-ACP. Mientras, KAS I y
II utiliza beta-cetoacil-ACP como la unidad de cebado, KAS III utiliza acetil-CoA. En la
mayoría de las plantas, los productos principales del ciclo de elongación de los ácidos
grasos son 16:0 y 18:0-ACP-ACP.
En Jatropha, un gran número de patógenos bacterianos, virales y fúngicos,
ocasiona considerables pérdidas de la producción sobre todo en postcosecha. Además,
de que posee una gran cantidad de ácidos grasos insaturados lo que ocasiona baja
calidad de aceite para convertirlo a biodiésel.
El análisis de un genoma involucra la identificación del gen que codifica para
proteínas y la asignación de la función probable a cada una de estas secuencias. El
programa BLAST (herramienta de búsqueda de secuencias similares por alineamiento)
busca secuencias homólogas en dos grandes bases de datos de secuencias: el GenBank
y el SWISS-PROT. La búsqueda usando BLAST está disponible en Internet a través del
Centro Nacional de Información Biotecnológica de los Estados Unidos (NCBI)
[http://www.ncbi.nlm.nih.gov/].

36
2.2.1. Alineamiento de secuencias de aminoácidos
La traducción a aminoácidos de las secuencias que codifican para las enzimas
ácido graso sintasa (KASII), reportadas en diversas especies, se analizaron para
comparar las similitudes de la enzima con otras especies vegetales, estas se utilizaron
mediante el programa BLAST (http://www.ncbi.nlm.nih.gov). Para el estudio tomó 1
secuencia completa de KASII publicada para Jatropha curcas depositada en el Gen Bank.
2.2.2. Diseño de oligonucleótidos
De acuerdo a los datos analizados en relación con la secuencia y estructura de la
enzima KASII se diseñaron oligonucleótidos específicos para amplificar la secuencia
completa, utilizando el programa Primer 3 Plus 2.3.5 (Jurka, J., et al., 1996.) con base a la
secuencia de KASII de J. curcas reportada en el Gen Bank (gb|DQ987700.2). Todos los
cebadores diseñados en este trabajo experimental, fueron sintetizados por SIGMA
ALDRICH.
2.2.3. Material vegetal de J. curcas
Se utilizaron diferentes tipos de tejidos vegetal: Hoja, tallo y frutos, como se
describe a continuación: para determinar la expresión basal en los diferentes días post-
antesis de los frutos de J. curcas, se utilizaron ARN de diferentes días de maduración (25,
30 y 35) colectadas de la plantación experimental del invernadero del CICY. Se usó tejido
proveniente de hoja verdadera y tallos de plántulas obtenidas in vitro. El material vegetal
se mantuvo en refrigeración a -80 °C hasta su procesamiento (Fig. 2.4).

37
Figura 2.4. Material vegetal de Jatropha curcas de A. Plantula in vitro. B. Frutos inmaduros. C.
Fruto con Semillas maduras.
2.2.4. Extracción de ARN.
a. El material biológico a utilizar fueron hojas y semillas de J. curcas (100 mg) y al
instante fueron congeladas en N2 líquido.
b. Se adicionó 1 mL de Trizol® Reagent (INVITROGEN Lot. #64901) para 50-100 mg
de tejido y homogenizar la solución.
c. Centrifugué a 12,000 x g por 10 min a 4 °C, y el sobrenadante, se transfirió a un
tubo nuevo.
d. Sometí el sobrenadante a temperatura ambiente por 5 min y se adicionó 0.2 mL
(200 µL) de cloroformo (sustituido por 2,3-propano) y agitó vigorosamente con
movimientos invertidos con la mano 15 veces.
e. Se almacenó por 2-3 min a temperatura ambiente y centrifugué a 12,000 x g por
15 minutos a 4 °C. Se removió la fase acuosa; inclinando 45° el tubo para extraer
la fase, procurando no llevar consigo extracto de la otra fase inmediata. Añadí la
fase a un tubo nuevo para aislar el ARN.

38
f. Posteriormente adicioné 0.5 ml (500 µL) de isopropanol al 100 % por 1 mL de
Trizol®; y mantuve a temperatura ambiente por 10 min o una hora.
g. Centrifugué 12, 000 x g por 10 min a 4 °C (Formación de pellet tipo gel).
h. Removí el sobrenadante, dejando solo el pellet de RNA en el fondo del tubo.
i. Posteriormente lavé el pellet con 1 mL (1000 µL) de etanol al 75 %.
j. En un vortex homogenizé la pastilla unos segundos, después centrifugué a 7500 x
g por 5 min a 4 °C y descarté el sobrenadante, dejando una vez más la pastilla en
el fondo del tubo.
k. Finalmente, coloqué de manera invertida, el tubo con el pellet en una sanita de
papel; aproximadamente 5-10 min para secar.
l. Una vez secado el pellet, resuspendí en agua libre de RNasa o DEPC de 10 a 30
µL. Utilizar o llevar a -80 °C.
m. Medí absorbancia A260/280 nm <1.6
n. Fraccioné por electroforesis-agarosa 1 %.
2.2.5. Síntesis de cDNA por RT-PCR
Para la obtención del ADN complementario [56] se siguió el protocolo de
SuperScriptTM First-Strand Synthesis System for RT-PCR (Invitrogen Cat No. 11904-
018).
Se realizó la siguiente mezcla en un tubo Eppendorf de 1.7 mL: Mezcla ARN: ARN
3 µg, dNTP mix 2.5 µM, Primer [0.5 µg / µL oligo(dt)], llevé a un volumen de 10 µL con
agua DEPC.
Una vez realizada la mezcla de RNA/primer esta se mantuvo a una temperatura de
65 °C por 5 min, posteriormente, se colocó en hielo por 1 minuto. En un tubo Eppendorf
adicional se preparó la mezcla 1 RX de reacción con los siguientes componentes en
orden: 10X RT buffer, 25 mM MgCl2, 0.1 DTT, RNasa Out TM (40 U / µL).

39
Una vez hecha la mezcla de reacción se adicionó 9 µL de volumen total para cada
tubo Eppendorf con la mezcla RNA/primer mezclando gentilmente y colectar la reacción
por centrifugado rápido, la reacción completa se incubó a 47 °C por 2 min, pasado el
tiempo de incubación se adicionó 1µL de la Super ScriptTM II RT de INVITROGEN para
cada tubo, esto se incuban a 42 °C por 50 min, la reacción se terminó incubando
nuevamente a 70 °C por 15 min y se colectó por centrifugado rápido, adicionando
posteriormente 1 µL de RNasa H para cada tubo manteniendo a una temperatura de 37
°C por 20 min. La reacción se almacenó a -20 °C para posteriormente utilizarse en la
reacción de PCR.
2.2.6. Condiciones de PCR
Para obtener el fragmento de las secuencias sintetizadas se utilizó la técnica de
PCR (reacción en cadena de la polimerasa), con la cual se amplificaron las secuencias de
ADN del gen KASII. Esta técnica, se basa en hibridar los oligos diseñados a partir
mediante la actividad de la enzima Taq polimerasa. Para amplificar un fragmento del gen
de esta enzima se utilizaron pares de cebadores especificos. De estos oligonucleótidos el
primer par contiene nucleótidos adicionales al inicio de la secuencia que codifican para un
sitio de restricción. También se establecieron las condiciones ideales de hibridación de
ADN, utilizando gradientes de temperatura (Fig. 2.3).
Se realizaron mezclas de reacción para PCR de 25 µl conteniendo 2.5 µl de
amortiguador 10X para PCR (Invitrogen), 2 µl MgCl2 (50 mM), 1 µl de dNTPs (10 mM), 1
µl de cada uno de los oligos Forward y Reverse (20 pm/µl), 15.5 µl de agua ultrapura y 1
µl de la enzima Taq polimerasa (1 U/pmol).
La reacción finalmente se llevó a cabo en un termociclador marca 5Prime,
mediante las siguientes condiciones: desnaturalización 95°C por 30 s, seguido de 35
ciclos a 95 °C por 30 s, 50-60 °C por 30 s y 72 °C por 2 min; y una extensión final de 72
°C durante 7 min.

40
Las muestras se fraccionaron en un gel de agarosa al 1%, teñido con gel red a 90
V durante 45 min.
Figura 2.3.Condiciones de amplificación para los diferentes pares de oligonucleoticos utilizados.
2.2.7. Análisis de expresión de JcKASII mediante qPCR
La PCR cuantitativa (qPCR o Q-PCR) o PCR en tiempo real, es una variante de
la reacción en cadena de la polimerasa (PCR) utilizada para amplificar y simultáneamente
cuantificar de forma absoluta el producto de la amplificación de ADN. Para ello emplea,
del mismo modo que la PCR convencional, un molde de ADN, al menos un par
de cebadores específicos, dNTPs, un buffer de reacción adecuado, y una ADN
polimerasa termoestable; a dicha mezcla se le adiciona una sustancia marcada con
un fluoróforo, que en un termociclador a la longitud de onda apropiada, permita medir la
tasa de generación de uno o más productos específicos.
Para esta técnica, se utilizó el siguiente Kit EXPRESS SYBR GreenER SUPERMix
with Premixed ROX™ de INVITROGEN Cat. No. 1167485. Utilizando el equipo Applied
Biosystems StepOne Real-Time PCR System, las condiciones de amplificación fueron las
siguientes: desnaturalización inicial 94 °C durante 3 min; desnaturalización: 35 ciclos de
94°C por 30 s, extensión 52 °C por 30 s y 72°C por 2 min; y una extensión final de 72°C

41
durante 7 min. Las muestras utilizadas fueron obtenidas de la obtención del ADNc en
diferentes días post-antesis.
2.2.8. Transformación de la cepa E. coli DH5-α
1. Tomé una alícuota (100 L) de bacterias competentes e incubé en hielo durante
15 minutos (descongelación lenta).
2. Añadí 1 ml (1ng) de plásmido (o producto de ligación).
3. Almacené en hielo durante 20 min, posteriormente incubé a 42°C durante 45 s.
4. Transferí a hielo e incubé durante 2 min.
5. Agregué 900 µl de medio LB e incubé 90 min a 37 °C en agitación a 200 rpm.
6. En cajas Petri previamente hechas con medio LB + Amp [100 mg/L], se agregaron
30 L de X-Gal [20 ng/mL], se esparció por la caja y esperamos a que secará.
7. Una vez que se ha secado el X-Gal, agregué 50 L de la suspensión bacteriana y
esparcí en toda la caja.
8. Finalmente se plaquearon 100 mL en medio selectivo los productos de ligación.
9. Incubé las cajas a 37 °C toda la noche (12 a 16 h) y observé la formación de
colonias.
Nota: Para plásmidos que presentan complementación, 2 l de IPTG de concentración
inicial 1 M y 25 a 30 L de X-Gal (solución stock: 20 mg de la droga sólida disuelta en 1
mL de N, N-dimetilformamida) por placa chica.

42
2.2.9. Protocolo de minipreparación de ADN plasmídico {Birnboim, 1997 31478 /id}
a. Centrifugué 2 ml de cultivo bacteriano de la noche anterior en un tubo Eppendorf®
a 6000 rpm a 4°C durante 1 min. Posteriormente se decantó el medio.
b. Resuspendí en 100 µL de amortiguador TEG (25 mM tris-HCl, 10 mM EDTA, 50
mM Glucosa) y dejé a T° ambiente por 5 min.
c. Agregué 200 µL de amortiguador NS (NaOH 0.2 M y 0.5% SDS) y homogenicé
delicadamente.
d. Dejé a T° ambiente durante 5 min.
e. Agregué 150 µL de amortiguador de acetato de potasio y se homogenizó
delicadamente.
f. Almacené 5 min en hielo, posteriormente centrifugué a 12500 rpm durante 20 min.,
luego recuperé el sobrenadante.
g. Agregué 700 µL de etanol absoluto e incubé desde 30 min o toda la noche a -20
°C.
h. Centrifugué durante15 min a 10,000 rpm y decanté sobrenadante.
i. Lavé pastilla por pipeteo con etanol al 70% dos veces y dejé secar a temperatura
ambiente o a 37 °C aproximadamente 30 min.
j. Resuspendí el ADN con 25 µL de agua + ARNasa.
k. Medí concentración y fraccioné por electroforesis en gel para comprobación de
integridad.
l. Almacené muestras a -20 °C.

43
2.3. Mapa de los plásmidos pCAMBIA2301, pGEM®-T Easy
Para la transformación de los callos utilicé el plásmido pCAMBIA2301 (Figura 2.5).
Figura 2.5. Mapa de restricción de pCAMBIA2301. Se observa el tamaño del plásmido (11, 633 pb),
el promotor constitutivo CAMV35S. Sitios de restricción de enzimas, gen de resistencia para
selección bacteriana, HYG (higromicina) y gen de resistencia para selección de plantas
transformadas Kanamicina.
Para la clonación del gen KASII, utilicé el vector pGEM®-T Easy (Figura 2.6).

44
Figura 2.6. Vector de clonación pGEM®-T Easy. Se observa el tamaño (3016 pb), el promotor T7,
Sitios de restricción de enzimas, gen de resistencia para selección bacteriana, AmpR (ampicilicina).
2.3.1. Protocolo para obtención de células competentes
Para obtener las células competentes se utilizó el siguiente amortiguador:
Buffer M: 15% de glicerol, CaCl2 75 mM y aforar con H2O bidestilada, esterilizar por 20
min a 121 °C.
Se utilizó medio LB (Luria–Bertani) liquido estéril, si las células a tratar presentan
resistencia a uno o más antibióticos, dichos antibióticos deberán estar presentes en el
medio en las concentraciones adecuadas para dichas células.
Piqué una colonia de células bacterianas e inoculé 5 mL de medio LB, incubé a 37
°C y agité durante toda la noche, transferí 2 mL del cultivo a 200 mL de medio LB estéril, e
incubé a temperatura y agitación controladas hasta alcanzar valores de OD600 de 4.0 +/-
0.5; dividir los 200 mL de cultivo en tubos de 50 mL estériles (previamente enfriados en
hielo), e incubé en hielo durante 5 min. Posteriormente se colectaron las células por
centrifugación a 1000 g durante 5 min a 4 °C y decanté el sobrenadante, y resuspendí las
bacterias en 20 mL de buffer M (previamente enfriado en hielo) a cada tubo de 50 mL y se
dejó incubar por 5 min en hielo, posteriormente se colectaron las células por

45
centrifugación a las mismas condiciones y se decantó el sobrenadante; nuevamente se
resuspendieron en 20 mL de buffer M y se incubó en hielo centrifugué y descarté el
sobrenadante. Finalmente, resuspendí las células en 4 mL de buffer M y realicé alícuotas
de 100 µL para ser congeladas a -80°C.
Posteriormente, se realizó la transformación de la cepa E. coli DH5α, con el
protocolo descrito anteriormente. Se picaron las colonias que crecieron en el medio solido
(YEB) y se suspendieron dentro de tubos cónicos en 5 mL de medio de crecimiento LB
líquido con el mismo antibiótico e igual concentración.
Finalmente, se verificó la introducción de los plásmidos pGEM y pCAMBIA1301, en
E. coli DH5α por digestión del ADN extraído de sus respectivas suspensiones mediante
una doble digestión con HindIII y EcoRI.
2.3.2. Preparación del cultivo de A. tumefaciens
Inoculé A. tumefaciens LBA 4404 (resguardo celular en glicerol a -80 ºC o una
colonia) en 5 mL de medio YEB, el cual contiene los antibióticos apropiados (100 mg·L-1
estreptomicina, 50 mg·L-1 rifampicina, 50 mg·L-1 kanamicina) y dejé bajo condiciones de
oscuridad a 28 ºC con agitación de 200 rpm durante 48 h. Posteriormente, se tomaron 250
µL de este cultivo bacteriano y se colocaron un matraz Erlenmeyer de 50 mL que contenía
25 mL de medio YEB suplementado con los antibióticos requeridos y acetosiringona a una
concentración final de 100 µM. El cultivo bacteriano se mantuvo a una temperatura de 28
ºC en oscuridad y agitación a 200 rpm hasta alcanzar una densidad óptica de OD600= 0.3-
0.5. Finalmente, centrifugué a 2500 g durante 10 min y resuspendí en 25 mL de medio
YEB o MS suplementado con acetosiringona a una concentración final de 200 µM y 0.05%
de Sweet L-77.

46
2.3.3. Preparación de células competentes de A. tumefaciens
i. Crecí una colonia de A. tumefaciens en 2 mL de medio YEB a 28 °C en oscuridad.
ii. Inoculé los 2 ml en 50 mL de medio YEB y dejé crecer hasta alcanzar una OD600
de 0.5.
iii. Centrifugué por 5,000 g por 5 min y descartar el sobrenadante.
iv. Resuspendí la pastilla en 10 mL de 0.15 M de NaCl.
Centrifugué por 5,000 g por 5 min, y resuspendí la pastilla en 1 mL de CaCl2 a 20
mM y 10% de: glicerol.
v. Distribuí alícuotas de 200 µL de células en tubos Eppendorf e introducí en N2
líquido para almacené a -80 °C.
2.3.4. Transformación de la cepa bacteriana A. tumefaciens
Se realizó por el método de congelación-descongelación reportado por Zhang y
Zeevaart (1999).
a. Tomé 1 µg de ADN plasmídico e inoculé en un tubo Eppendorf que contiene 200 µL
de células competentes de A. tumefaciens LBA4404, se almacenaron por 30 min en
hielo. Posteriormente la solución se introdujo en nitrógeno líquido durante un minuto,
y después se descongeló a 37 ºC.
b. Posteriormente, se adicionó 1 mL de medio YEB (5 g·L-1 extracto de carne, 1 g·L-1
extracto de levadura, 5 g·L-1 peptona, 2 mM sulfato de magnesio, pH 7.2) y se
mantuvo a 28 ºC durante 4 h con agitación a 200 rpm, a continuación se centrifugó a
14,000 g por un minuto y se descartó el sobrenadante, la pastilla se resuspendió con
100 µL de medio YEB.
c. A continuación, se inocularon las cajas de Petri que contienen medio YEB sólido con
los antibióticos adecuados (100 mg·L-1 estreptomicina, 50 mg·L-1 rifampicina, 50
mg·L-1 kanamicina). Se seleccionaron colonias positivas para inocular en tubos de
ensayo con 6 mL de medio YEB con antibióticos, finalmente se mantuvieron durante
48 h a 28 °C bajo condiciones de oscuridad a 200 rpm.

47
2.3.5. Transformación mediante Agrobacterium en tejidos in vitro de J. curcas
Cultivé A. tumefaciens (50 mL) e inoculé con 500 µL a una densidad óptica de 1 a
600 nm.
Posteriormente coseché por centrifugación (4000 g por 8 min).
Resuspendí las células en medio MS+Vit. B5 (sin RCV) + Acetosiringona a 200
µM.
Se mantuvo a una temperatura de 28 °C durante 2 h a 200 rpm.
Coloqué los explantes (callos) en la solución de infección (cepa bacteriana +
acetosiringon y dejé bajo condiciones de oscuridad a una temperatura de 28 °C
durante 1 h a 200 rpm.
Transferí los callos a medio liquido (MS+ Vit. B5) suplementado con acetosiringona
(cambiar a nuevo matraz de 50 ml) a 28 °C en condiciones de oscuridad a 200 rpm
durante 72 h.
Finalmente, coloqué los callos en antibiótico (Cefotaxima 400 mg L-1) durante 72 h
suplementado con medio MS+BA+AIA.
Transcurrido el tiempo realizar prueba de GUS.
*la prueba de GUS se puede hacer con dos sustratos distintos: 5-bromo-4-chloro-3-indolyl
glucuronide (X-Gluc) Biosynth + 20% v/v de metanol (inhibe la actividad endógena de
GUS) y usando 4-methylumbelliferyl β-D-glucuronide (4-MUG), la prueba con este
sustrato no destruye los explantes y se pueden seguir propagando.
2.3.6. Localización histoquímica de GUS en tejidos in vitro de Jatropha
Se colocaron callos de Jatropha en un tubo Eppendorf de 1.5 mL (protegieron de
la luz).
Adicioné la solución sustrato X-Gluc con el amortiguador (1 mg mL); antes de
añadir ajusté el pH a 7 (Tab. 2.1).
Mantuve las muestras a una temperatura de 37 °C durante 1 h.

48
Para desteñir el tejido utilicé metanol: acetona (3:1) dos repeticiones de 24 h.
Por último embebí el tejido en glicerol: agua (1:1).
Se observaron al estereoscopio y se documentaron con fotografías.
Tabla 2.1. Componentes del amortiguador de X-GlcA
2.4. Obtención del material vegetal in vitro
Las semillas de J. curcas fueron recolectadas en el invernadero dentro de las
instalaciones del CICY. La recolección de semillas se realizó dependiendo de la madurez
del fruto, para obtener las plántulas in vitro necesarias para los respectivos experimentos.
Posteriormente se desinfectaron las semillas de acuerdo al siguiente protocolo:
Material:
Extrán 5%
Agitador magnético
Agua estéril
Frascos de 500 mL
Etanol 70%
Hipoclorito (cloralex®)
Componente Concentración final
Fosfato de sodio pH 7 100 mM
EDTA pH 8 10 mM
Ferricianuro de potasio 0.5 mM
Ferrocianuro de potasio 0.5 mM
Triton X-100 0.1%
X-Gluc 2 mM

49
PPM 2%
Bisturí
Campana de flujo laminar
Procedimiento
I. Lavado
Las semillas recolectadas fueron lavadas con agua y jabón por fuera y se mantuvo
en agitación durante un intervalo de 10 minutos. Al terminar, se eliminó el exceso de jabón
enjuagando tres veces con agua destilada, inmediatamente se pusieron a secar, para
posteriormente retirar la testa. Posterior al retiro de la testa, se colocaron en un matraz
con agua destilada para llevar a cabo la desinfección en las campanas de flujo laminar.
II. Desinfección
En la campana de flujo laminar, se preparó una solución de Extrán al 5% [32] para
lavar las semillas durante cinco minutos en agitación. Se decantó la solución y se lavaron
las semillas tres veces con agua estéril para quitar el exceso de Extrán. Después se
dejaron agitando en una solución de etanol al 70% [32] etanol por un periodo de 3
minutos, se decantó la solución y las semillas se lavaron 3 veces con agua estéril.
Posteriormente, las semillas se agitaron durante 15 minutos en una solución de hipoclorito
de sodio al 10% [32], las semillas se lavaron cinco veces con agua estéril, para quitar el
exceso de hipoclorito de sodio. Finalmente, las semillas se dejaron reposar por ocho
horas en una solución de Preservative Plant Medium (PPM) al 2% [32], después de este
tratamiento, están listas para proceder a la extracción de los embriones cigóticos.
III. Germinación
La extracción de los embriones cigóticos se realizó en condiciones asépticas, en
una campana de flujo laminar, con un bisturí, abriendo el endospermo de forma
longitudinal. Con unas pinzas se tomó el embrión cigótico y se sembró en el medio de
germinación. Inicialmente se sembraron 20 embriones cigóticos. El medio de cultivo MS

50
(1962) fue suplementado con sacarosa cisteína, mio-inositol, tiamina y PPM (Tabla 2.2).
El pH del medio se ajustó a 5.8 y se adicionó gel Rite. La esterilización del medio de
germinación se llevó a cabo en autoclave por un periodo de 20 minutos a una temperatura
de 120° C. Los embriones cigóticos fueron colocados en cajas magentas en las que
fueron cultivados en condiciones de fotoperiodo (16/8 h luz-oscuridad a 25 °C), hasta su
regeneración de plántulas para la obtención de las primeras hojas verdaderas con una
edad aproximadamente 21 días, y se mantuvieron hasta su transformación.
Tabla 2.2. Composición de medio MS para germinación.
MS 4.3 g/lL
Sacarosa 30 g/L
Mio-inositol 100 mg/L
Cisteína 25 mg/L
Tiamina 4mg/L
Gel rite 2.3g/L
2.4.1. Inducción de callo
Para la inducción de callos se repitieron tratamientos con diferentes
concentraciones de auxinas y citocininas en condiciones de fotoperíodo (16 h luz-/8 h
oscuridad, 25º C). El experimento consistió en utilizar explantes de hoja, hipocotilo y
plántula de J. curcas cultivadas in vitro, se probaron 2 tratamientos con la finalidad de
determinar cuál es la concentración de RCV (auxinas y citocininas) y con qué tipo de
explante se tenía una mejor respuesta a la inducción de callo. Se mantuvieron en cada
medio de inducción hasta su transformación (Fig. 2.7).

51
Figura 2.7. Protocolo de inducción para J. curcas
Tabla 2.3. Medio de inducción de callos (IC)
Reactivo Cantidad g/L
Sales MS 4.3
Mioinositol 0.1
Acido nicotínico 0.001
Tiamina-HCL 0.01
Piridoxina-HCL
Sacarosa 30
*AIA 0.010
*BA 0.005
PPM 2%
pH= 5.8

52
Tabla 2.4. Medio de inducción de brotes (IB).
Reactivo Cantidad g/L
Sales MS 4.3
Mioinositol 0.1
Acido nicotínico 0.001
Tiamina-HCL 0.01
Piridoxina-HCL
Agar 8
Sacarosa
IBA
KIN
30
0.059
0.027
PPM 2%
pH= 5.8

53

54
CAPÍTULO III
RESULTADOS Y DISCUSIÓN
3.1. INTRODUCCIÓN
La sobreexpresión de un gen, se describe como el uso de un promotor fuerte con
la finalidad de inducir la transcripción constitutiva de la secuencia codificante del gen, el
efecto final es conseguir niveles altos y estables de proteína [16]. Dentro de las
aplicaciones importantes de la sobreexpresión de genes, se encuentran: la producción
aumentada de metabolitos secundarios y la generación de fenotipos tolerantes al estrés
abiótico y biótico. En un estudio reportado en el 2012, la sobreexpresión del ADNc en
JcKASII con un promotor de 35S dentro del virus del mosaico de la coliflor en Arabidopsis
thaliana, resultó en un decremento del ácido palmítico y un incremento del ácido estéarico
tanto en hoja como semillas.
3.2. Alineamiento de KASII.
El objetivo de esta parte, fue determinar el número de genes que codifican para
KAS en la especie de Jatropha. Para ello se realizó una búsqueda en la base de datos del
genoma secuenciado de J. curcas por nombre [57].
En la tabla 3.1 se enlistan las especies para las cuales se obtuvieron secuencias
completas para KASII, así como sus correspondientes números de accesión. Estas fueron
alineadas usando el programa CLC Sequence Viewer 6.
Tabla 3.1. Números de accesión de las secuencias de aminoácidos de las KASII
seleccionadas para este estudio.
Especie No. accesión Jatropha curcas Q000L2
Ricinus cumunis Q41134
A. thaliana NP565097
Cuphea wrightii P93117
Glycine max Q5ECI5

55
La búsqueda in silico de secuencias que codifican para KASII en J. curcas, dio
como resultados un sólo gen (Tab. 3.2).
Con la secuencia de proteínas de KASII se procedió a compararla con la
secuencia de las otras cuatro especies listadas en la tabla 3.1. El resultado del
alineamiento de las secuencias de la traducción a proteína mostró un alto porcentaje de
identidad con las secuencias de otras especies (Fig. 3.1). El sitio activo para la proteína
de KASII (Cys 324) no produjo cambios.
Tabla 3.2. Genes involucrados en la síntesis de ácidos grasos.
Nombre del gen
Genes en A.
thaliana
Jatropha conting./ Jatropha genes
Jatropha ARNm depositado en
genbank
R. comunis genoma modelo
3-cetoacil-ACP sintasa (EC.2.3.1.41)
KASI At5g46290 JCS201881 (JcCB0272931.20, JcCB0282211.10)
DQ987699.1 XP_002519707.1
KASII At 1g74960 JCS200398 (JcCA0028141.10, JcCA0143871.10)
DQ987700
XP_002516228.1
KASIII At1g62640 JCS100375 (JcCB0393911.10, JcCD0062224.10, JcCB0043371.20, JcCB0199771.10,
DQ987701
XP_002529789.1

56
Figura 3.1. Alineamiento de secuencias para KASII de J. curcas con proteínas de otras especies.
Los aminoácidos marcados en el mismo color indican la identidad. El sitio activo Cys324 se indica
con flecha vertical color azul.
3.2.1. Diseño de oligonucleótidos
Con base a los datos analizados y a lo reportado en la literatura, se decidió
diseñar un oligonucleótido específico para obtener el ADNc que corresponde a KASII con
un tamaño de 1712 pb como se muestra en la figura 3.1 las flechas marcadas en color
morado indican la región inicial y la final de la secuencia 5´y 3´.

57
Los oligonucleótidos utilizados para el presente proyecto se muestran en la tabla
2.3 de los cuales el PAR1 corresponde a actina, PAR2 un pequeño fragmento de KASII,
estos pares fueron utilizados como testigo (diseñados por Cristy Catzin); PAR3
corresponde al oligonucleótido diseñado para atrapar el gen completo; PAR 4 y 5
corresponden a una región del gen de KASII obtenidos de la literatura reportada.
Figura 3.2. Secuencia completa del gen KASII y diseño de oligonucleótidos. Las flechas indican el
plegamiento de los cebadores diseñados (5´ - 3´).

58
Tabla 3.3. Secuencias de oligonucleótidos y longitud de amplicones correspondientes (5´-
3´).
El alineamiento nos permitió obtener una identidad del 85% para KASII en
Jatropha con otras plantas. El sitio activo se conserva para todas las especies. El marco
de lectura abierto corresponde a 573 aminoácidos, con un residuo conservado Cys 324, el
cual pertenece al sitio activo de la proteína KASII.
Los oligonucleótidos se diseñaron con base a lo reportado en la literatura y se
tomaron como base los cebadores PAR4 y PAR5, los cuales pertenecen a una región del
gen, ya que con esta región pudieron sobre expresar, no obstante nos recomendaron
contar con la cantidad posible del gen, si era posible completo era mucho mayor el
resultado y confiable a nivel biológico.
Dichos cebadores como se muestra en la figura anterior, se diseñaron en regiones
específicas y aunque algunas pares de bases sobran, ya que al momento de realizar el
diseño el cebador 5´producía errores, por ello se decidió mover la región del 3´para
obtener de esta forma la secuencia completa.
Cebador Secuencia Long. del Amplicón
Par 1 JcActina F: ATGAGCTTCGAGTTGCACCA
R:AGCATCAGTGAGATCACGAC
200 pb
Par 2 JcK2 F:GAAGAAACCAGCTACGAAGC
R: AGTGATACCACCATCAGCCA
289 pb
Par 3 KASII F: GATTCTAGATGATGGGGTCAGCTTCGT
R: GATAAGCTGGCGCAAAAACGATTGAT
1722 pb
Par 4 KASII
Par 5 KASII
F:TCGAGCATTTGTATCCACGCCTTCG
R:TGCTCTAGATTTCTCGATTGACATTTTCT
F: CTAAATGTGGAGTTTTGATTGGCT
R:ACAGGCAGTGGAGATTGAATAGTTT
1310 pb
1500 pb

59
3.3. Aislamiento de ARN de diferentes tejidos de J. curcas
El ARN fue aislado a partir de tallos, hojas verdaderas y frutos maduros e
inmaduros, sin degradación ni contaminación. Para los frutos se colectaron diferentes días
25, 30 y 35. En el gel de agarosa se observaron el par de bandas correspondientes a las
subunidades 25 S y 18 S. Las bandas obtenidas en semillas expresaron mayor contenido
de expresión, sin embargo, en los tejidos de hoja y tallo no pudo aislarse ARN (Fig. 3.3).
Figura 3.3. Gel de agarosa con muestras de ARN en diferentes tejidos vegetales; H. Hojas, T tallo,
Semilla en diferentes días post-antesis (dpa).
El mayor rendimiento promedio de ARN obtenido por tejido, se obtuvo en semillas,
en particular del día 25 y 35; por el contrario el de más bajo contenido en el día 30 y el de
tallos y hoja (Tab. 3.4).
Tabla 3.4. Contenido de ARN de las muestras vegetales para el estudio.
Tejido A 260/280 [µg ARN 100-1
mg-1
tejido fresco]
Hoja 1.48 0.1048
Tallo 1.25 0.348
Semillas
D25 2.06 2.3985
D30 1.99 0.9922
D35 1.98 0.7002

60
3.3.1. Análisis de expresión de KASII mediante qPCR
Los cálculos en cuantificación relativa de expresión genética se basan en la
comparación de los valores Ct utilizando la eficiencia de la reacción de la PCR como
factor de corrección. Sin embargo, hay un modelo que no requiere la eficiencia de la
reacción para acceder a un factor de corrección. Este modelo supone una eficiencia
óptima e idéntica (correspondiente al 100%) en la eficiencia de reacción en las PCR en
tiempo real tanto del gen en estudio como del gen de referencia (Livak & Schmittgen,
2001). Este es el método 2 delta-delta Ct que sólo es aplicable para una estimación rápida
de la proporción relativa de la expresión genética en estudio. El método 2 delta-delta Ct
expresa la proporción obtenida de la relación entre los valores Ct de la muestra y los
valores Ct del control tal y patológicas o experimentales, así como para la cuantificación
de niveles.
Para la detección de cambios cuantitativos en la expresión génica de JcKASII
durante el curso de las etapas de los frutos, mostraron que JcKASII se encuentra
presente en todos los dpa examinados y aumentando el contenido de expresión a los 30
dpa. Sin embargo, durante la última etapa de maduración se pudo ver un decremento en
el nivel de expresión (40 dpa) (Fig. 3.4).

61
Figura 3.4. Analisis de expresion cuantitativa de KASII en los diferentes dias post-antesis. El nivel
de expresion fue mayor a los 30 dpa. Los niveles de expresion fueron estandarizados con la
JcActina, para cada muestra se utilizaron tres replicas.
3.3.2. Patrón de expresión del gen KASII en diferentes días post-antesis de frutos
EL ADNc obtenido y usado como plantilla para la amplificación por PCR de punto
final con los oligonucleótidos específicos para el gen constitutivo ActinaJc, generó
amplicones de diferente densidad (Fig. 3.5).
El gen de ActinaJc presentó bandas uniformemente intensas en diferentes días
post-antesis.
Figura 3.5. Amplificación del transcrito ActinaJc con 230 pb de J. curcas, a partir de 3 µg de ARN.
Se tomó 1 µL de ADNc. Se cargó 10 µL de producto de PCR. Gel de agarosa al 1 % y teñido con
Gel red. T. Tallo, H: Hoja, Días post-antesis.

62
PAR2-KASII, estos cebadores sirvieron como testigo del gen JcKASII con un
amplicón de 239 pb. Se observó una banda intensa en fruto maduro correspondiente al
dia 30 post-antesis (ver Fig. 3.6).
Figura 3.6. Gel de agarosa en donde se observa la expresión de JcKASII en semillas de maduras
(30 dpa) obtenidos con los cebadores PAR2 de un tamaño de 239 pb.
PAR3-KASII generó bandas inespecíficas a los 35 días post-antesis, por lo cual se
decidió realizar nuevamente la reacción de PCR en los mismos días y realizando diversas
condiciones de PCR, pero después de varios intentos logrando buscar el amplicon
deseado no se observó ninguna banda; se decidió probar los cebadores siguientes para el
mismo gen (Fig. 3.7).
Figura 3.7. Amplificación del transcrito de JcKASII con los cebadores PAR3, obtenido a los 35 dpa.
Se cargó 10 µL de producto de PCR. Gel de agarosa al 1 % y teñido con Gel red. MM: Marcador de
1 Kb. 1: 72, 2:71.1, 3: 67.3, 4:64.4, 5:62.3 y 6:60°C.

63
Para establecer el protocolo se utilizaron diversos oligonucleótidos mencionados
en el capitulo anterior, como se detalla a continuación:
PAR4-KASII, con estos cebadores se utilizaron las mismas condiciones para
estandarizar el protocolo de amplificación. Con estos oligonucleótidos se generaron varias
bandas inespecíficas de leves a intensas en los diferentes días post-antesis (Fig. 3.8).
Figura 3.8. Expresión de JcKASII con cebadores del PAR4 aprox. con 1312 pb. Se observan
bandas inespecíficas en los carriles a diferentes gradientes de temperatura, se pudo amplificar un
tamaño aprox de 1000 y de 1300 pb.
Sin embargo, se volvió a repetir el mismo procedimiento ahora en algunos días
post-antesis: 25, 30 y 35, generando bandas inespecíficas de igual manera (Fig. 3.9).

64
Figura 3.9. Gel de agarosa donde se puede observar la amplificación del transcrito de JcKASII
generados con PAR 4, a los 25, 30 y 35 dpa. Se cargó 10 µL de producto de PCR. Gel de agarosa
al 1 % y teñido con Gel red. MM: Marcador de 1 Kb.
Se decidió realizar un experimento de comprobación de integridad de cebadores
diseñados para lo cual se realizó una serie de combinaciones del PAR2 Y PAR3,
colocando en la reacción de PCR el foward y el reverse de cada uno (Fig. 3.10).
Figura 3.10. A. Combinación de cebadores C1 (R/F) PAR2 Y PAR 3; CASO 2 (F/R) PAR 3 Y PAR
2, A 50, 55 Y 60°C Día 25 post-antesis. B. Combinación de cebadores C1 (R/F) PAR2 Y PAR 3;
CASO 2 (F/R) PAR 3 Y PAR 2, A 50, 55 Y 60°C Día 30 post-antesis; se utilizó ActinaJc como
testigo.

65
No obstante, no se podía aislar la secuencia del ADNc para JcKASII, se
examinaron los cebadores PAR4 nuevamente pero ahora modificando las condiciones de
PCR, además de generar un nuevo ADNc del día 25 post-antesis (ver Fig. 3.11).
Figura 3.11. Expresión de JcKASII con cebadores del PAR3 y PAR 4 con un tamaño esperado de
1722 y 1312 pb respectivamente. Se observan bandas inespecíficas en los carriles a diferentes
gradientes de temperatura con un tamaño aprox de 1000 y 1600 pb para PAR3 y de 1300 pb para
el PAR 4. Se utilizó como gen constitutivo ActinaJc con un tamaño de 230 pb.
Con los cebadores PAR5 se siguió el mismo procedimiento anterior, pero no se
pudo obtener el tamaño esperado (1700 pb), aun con el gradiente de temperatura
utilizado, solo se pudo alcanzar un tamaño de 1000 pb, aunque con una leve expresión de
transcrito (Fig. 3.12).

66
Figura 3.12. Gel de agarosa donde se observa la expresión de JcKASII utilizando cebadores del
PAR5 aprox. con 1700 pb, bandas inespecíficas debido a los gradientes de temperatura,
amplificando un tamaño de 1000 pb. Se usaron testigos positivos como PAR 2 (JcKASII de 289 pb)
y PAR1 (ActinaJc de 200 pb) y el testigo negativo.
Obteniendo los resultados anteriores con PAR3 (diseñado específicamente para
obtener secuencia completa), se decidió realizar un segundo experimento, ahora con un
ADNc del día 30 y utilizando temperaturas de 50 a 60 °C, durante el cual se probaron las
condiciones mostradas en el capitulo anterior y utilizando una Taq Polimerasa casera, sin
embargo, no hubo expresión alguna (Fig. 3.13).

67
Figura 3.13. Amplificación del transcrito de KASII de J. curcas a los 30 dpa. Se cargó 10 µL de
producto de PCR. Gel de agarosa al 1 % y teñido con Gel red. MM: Marcador de 1 Kb. 1: 50, 2:52,
3: 55.3, 4:55.6, 5:60 y 6:60.8 °C.
Sin embargo, se encontraron varias dificultades que fueron remediadas
descartando cada uno de los problemas que se produjeron al paso de la investigación,
como el de la amplificación del transcrito de JcKASII, donde se pudo observar que se
necesita una serie de condiciones específicas como el tipo de Taq Polimerasa para
obtener un amplicon de esta longitud, en este caso se utilzó la LongAmp® Taq DNA
Polymerase M0323S (New England BioLabs®) con su respectivo Buffer y condiciones
necesarias según el instructivo. Además, de un ADNc íntegro y de buena calidad
descartando por supuesto los diferentes días de maduración del fruto (Fig. 3.14).

68
Figura 3.14. Gel de agarosa de la expresion del ADNc del gen JcKASII con los cebadores
especificos (PAR3) de un tamaño de 1722 pb. La reacción fue llevada a cabo con el ADNc del dia
25 dpa, con condiciones de amplificacion descritas en el manual de la Taq y utilizando 3
temperaturas de alineamiento; además de testigos (PAR 1 ActinaJc y KASIIJc de 289 pb).
Comparando los resultados con los reportados en el 2012 [58], durante la
expresión basal de JcKASII en diversos días de maduración del fruto de Jatropha, se
obtiene un resultado similar, donde JcKASII se expresa mayormente en frutos maduros.
Sin embargo, se encontraron dificultades para aislar la secuencia completa del
JcKASII, como el tipo de Taq Polimerasa para obtener un amplicon de una longitud de
casi 2000 pb; además, de las condiciones necesarias para la amplificación, etc.,
comparado con lo reportado en el 2012, donde el aislamiento del ADNc de la secuencia
completa fue realizado por la técnica de 3´ RACE, nosotros logramos obtenerlo mediante
una PCR convencional, descartando varias opciones.

69
3.3. Clonación del gen JcKASII en el vector pGEM ®- T Easy
Ya aislado el ADNc de JcKASII, se procedió a realizar la clonación en el vector
pGEM®-T Easy, y posteriormente, se envió a secuenciar y se comprobó que la secuencia
generada fuera la correcta, analizando un alineamiento de la secuencia clonada con la
secuencia depositada en el GenBank en el 2012.
Purificación de banda de 1722 pb (Kit Zymoclean Research DNA)
Con la ayuda de un bisturí corté la banda especifica, procurando un corte delgado,
posteriormente coloque en un tubo Eppendorf y procedí a pesar.
Añadí 3 volúmenes de ABD Buffer por cada 100 mg de gel de agarosa.
Mantuve a una temperatura de 55°C de 5 a 10 min, hasta fundir por completo la
agarosa.
Añadí la solución de agarosa y colecté en una columna zymo spin y un tubo
debajo de la misma.
Posteriormente, centrifugué durante 1 min y agregué 200 µL de Wash Buffer a la
columna y volví a centrifugar durante 1 min (repetí dos veces).
Pasé la columna a un tubo nuevo y agregué de 6-10 µL de agua tibia o
amortiguador de elución de ADN directamente a la columna matrix y centrifugué
para elucidar el ADN.
Por último comprobé la integridad en un gel de agarosa al 1 % (Fig. 3.15).

70
Figura 3.15. Producto de PCR purificado a diferentes diluciones de muestra.
Posteriormente, se realizó la ligación del producto al vector de pGEM®-T Easy
Vector System I (Promega Cat. No. A1360) de acuerdo al siguiente volumen de reacción
como se muestra en la tabla 3.4.
Tabla 3.4. Volumen de reacción utilizada en la ligación.
Reactivos 11 µL
Vector pGEM® 1
Inserto (KASII)
4
Buffer 5
Ligasa T4 1
Posterior a la ligación, se almacenó durante 1 h a temperatura ambiente, para
luego iniciar el proceso de transformación de E. coli DH5α con dicha ligación con el
procedimiento descrito anteriormente.
Seleccioné las colonias que resultaron positivas (blancas) las cuales crecieron en
el medio sólido (LB + Amp) y se suspendieron dentro de tubos cónicos en 5 mL de medio
de crecimiento LB líquido con el mismo antibiótico e igual concentración.
Posteriormente, se realizaron Minipreps para obtener el ADN plasmídico (Tab. 3.5)
se utilizaron los productos generados con los cebadores de PAR3 JcKASII con un tamaño

71
de 1722 pb y el generado por el cebador PAR5 de un tamaño de 800 pb
aproximadamente, durante la clonación en pGEM®-T Easy (Fig. 3.15).
Tabla 3.5. Concentracion de ADN genómico en las clonas que se obtuvieron
Clona [ng/µl] A260/280 A260/230
1 443.8 2.08 2.33
2 602.7 2.10 1.93
3 602.2 2.07 2.29
4 576.9 2.09 2.03
5 588.1 2.08 2.04
6 1371.8 1.68 1.02
7 625.2 2.09 1.92
8 621.8 2.07 2.23
9 763.7 2.10 2.33
10 415.1 2.11 1.82
11 480.1 1.97 2.37
12 499.1 2.03 1.80
13 858.2 2.09 2.22
14 863.4 2.11 1.90
15 344.2 2.09 2.10
16 201.7 2.09 1.78
17 353.0 2.04 1.51
18 236.5 2.04 2.07
19 446.1 2.01 2.43
20 410.0 2.09 1.97

72
Figura 3.16. Gel de Agarosa donde se observa la integridad del ADN plasmídico de las clonas
seleccionadas. Se cargó 2 µL de ADNg en el gel teñido con Red Gel ®. Los números azules
indican el número correspondiente a cada clona.
Finalmente, se verificó la introducción de pGEM® con JcKASII en E. coli DH5α por
medio de una PCR utilizando los cebadores específicos para cada clona correspondiente
(Fig. 3.17).
Figura 3.17. Electroforesis en gel del resultado de la PCR realzada a las colonias seleccionadas
usando cebadores específicos PAR3 y PAR5. Se observa el fragmento esperado (1722 pb) para
las clonas de la 6 a la 10 y de la 12 a la 16. Los números rojos indican el numero de clona
correspondiente.

73
Una vez obtenidas las clonas positivas, se enviaron muestras para su
secuenciación (clonas 6, 7, 9 y 12, 13 y 16) y verificar que el tamaño obtenido
corresponde al gen JcKASII.
El proceso de secuenciación se realizó, en la compañía CUGI Sequence,
solicitando la síntesis del oligo T7 y Sp6 específico para pGEM®-T Easy.
3.3.1. Análisis de de la secuenciación
Se realizó la búsqueda y comparación en la base de datos de la NCBI de la
secuencia de la clona 16. El BLAST nos proporcionó la siguiente información:
Tabla 3.6. Porcentaje de identidad comparado con otras especies de la secuencia
obtenida
Con este BLAST pudimos corroborar la identidad de nuestra secuencia obtenida
con la reportada en la base de datos obteniendo una identidad del 97% (Tab. 3.6).

74
Además de ello, se realizó un alineamiento con la secuencia reportada en el Gen
Bank utilizando dos programas el Snap Gene y el CLC Sequence Viewer 6.8.2 (Fig. 3.18 y
3.19).
Figura 3.18. Alineamiento de secuencias generadas para JcKASII. Las flechas rojas Derecha a
izquierda (5´a 3´) indican la secuencia que obtuvimos, ademas del empalmamiento de los
oligonucleotidos y la enzima de restricción HindIII; Flecha negra corresponde al fragmento
completo para KASII reportado en el GenBank de 1722 pb.

75
Figura 3.19. Alineamiento de la secuencia correspondiente al ADNc de JcKASII comparada con la
depositada en el Gen Bank.

76
Existen muchos promotores caracterizados que se utilizan en la transformación de
plantas, la elección de un promotor en particular depende del objetivo del proyecto de
transformación.
Hasta ahora, se han estudiado algunos de los genes que codifican para las
enzimas involucradas en la biosíntesis de los triacilglicéridos en J. curcas. Sin embargo,
no hay un estudio sistemático de todo el proceso, y que incluya la medida de las
actividades enzimáticas. Este es un campo de la investigación en J. curcas que requiere
de atención inmediata, por lo cual, este estudio realizado en esta enzima (KASII), nos
llevará a resolver muchas cuestiones y permitirá la introducción de esta en un vector de
clonación y de esta forma generar una variante transgénica, produciendo un posible
aumento en el contenido de ácidos grasos en el aceite de Jatropha.
3.4. Cultivo de cepas de A. tumefaciens
Para el experimento se utilizó una cepa comercial LBA4404 las cuales fueron
cultivadas y activadas de acuerdo al siguiente procedimiento (Fig. 3.20):
Preparación de medio LB estéril;
Agregué de 20-25 ml de medio en cajas petri.
Inoculé con una asa para bacterias y cultivadas en agar (YEB);
Colocar en orbitador de 28°C durante 48 h;
Posteriormente agregar el antibiótico.

77
Figura 3.20. Cultivo y activación de cepas de A. tumefaciens.
3.4.1. Efecto de los antibióticos en el crecimiento/resistencia de la bacteria
Para evaluar el efecto de los antibióticos en la cepa de A. tumefaciens se utilizaron
los siguientes antibióticos a diferentes concentraciones como se muestra en la tabla 3.7.
Tabla 3.7. Listado de antibióticos utilizados en la transformación.
Antibiótico Concentración Característica
Kanamicina 50 mg/mL Gen de resistencia en plásmido Ti
*Estreptomicina 0.5 g/mL Resistencia natural a la cepa 4404
*Rifampicina 50 mg/mL Cromosoma
Cefotaxima 0.33 g/mL Resistencia natural a la cepa 4404

78
Antes de utilizar los antibióticos se prepararon alícuotas para evaluar su efecto en
el crecimiento de la bacteria (Fig. 3.21).
Figura 3.21. Preparación de antibióticos y medio YEB para bacteria.
Se examinó el efecto de distintos antibióticos en el control y eliminación de A.
tumefaciens. Los antibióticos se utilizaron por separado o combinados entre ellos como se
muestra en la tabla 3.8.
Tabla 3.8. Antibióticos en diferentes combinaciones para de A. tumefaciens.
Combinación de
antibiótico
Concentración Terminología
Testigo S/A Medio YEP Sin antibiótico
0 10 µL cepa+ 20 ml YEP
YEP+ Bacteria
100 5 µL Es/ 20 µL Rf
Estreptomicina + rifampicina
50 (1) 5 µL Es Estreptomicina
50 (2) 20 µL Rf Rifampicina
Dos tubos c/u
+ 10 µL Cepa LBA4404
48 h en agitación

79
3.5. Germinación de embriones cigóticos
Las semillas de frutos maduros que se recolectaron fueron 20, en los invernaderos
del CICY. Posteriormente, se lavaron y esterilizaron para luego extraer los embriones,
según capítulo II. Estos fueron colocados en medio MS (1962) y se incubaron bajo
condiciones de fotoperiodo, los cuales se monitorearon en el transcurso del crecimiento
(Fig. 3.22).
Figura 3.22. Ciclo de obtención de embriones cigóticos de J. curcas.1. Obtencion de frutos y lavado
de semillas; 2. Desinfección en condiciones de asepxia; 3. Extracion del embrion cigótico y 4.
Germinacion en medio MS.
Se realizó un seguimiento del proceso de formación de las primeras hojas
cotiledonares de la plántula y es notorio el despegue de los cotiledonares para dar origen
a las demás hojas verdaderas en los días primeros 7 días.
Como es de esperarse, con el paso de los días las plántulas siguen alargando sus
raíces y se hacen más delgadas y frágiles. Alrededor de los días 15 y 18 disminuye la
velocidad de elongación del hipocotilo, debido a que la planta se está preparando para el
desarrollo de su primer par de hojas verdaderas. A continuación se muestra la descripción
de los eventos ocurridos durante la germinación de semillas de J. curcas (Fig. 3.23).

80
Figura 3. 23. Proceso general del desarrollo de los embriones para obtención de las plántulas in
vitro. A. Germinación de embrion cigótico; B. Día 2. C y D. Día 4 Enloganción de tallo. E-G. Día 7.
Abertura de los cotiledones. H-I. Día 10. Curvatura del tallo.
3.5.1. Inducción de callo
Posterior a la germinación, se esperó a la formación de las primeras hojas
verdaderas, es hasta el día 16, cuando se originan las primeras hojas verdaderas.
Después del día 30 las primeras hojas verdaderas están listas para ser utilizadas como
explante para la inducción de callos.
El segundo paso fue la obtención de callos a partir de explantes foliares de J.
curcas. Para ello se probaron 2 tratamientos a diferentes concentraciones de reguladores
del crecimiento (ANA, KIN, AIA, BA) y de tipos de explante (hipocotilo, hoja, plántula
completa).

81
En las figuras 3.24 y 3.25 se muestra el comportamiento de los explantes en los 2
tratamientos utilizados para la inducción de callo. El tratamiento uno (T1= ANA 2.69 μM,
KIN 4.64 μM + vitaminas MS) y el tratamiento 2 (T2= AIA 17.1 μM, BA 13.3 μM +
vitaminas B5) en condiciones de oscuridad tuvieron la mejor respuesta en la inducción.
Fue el T1 el que mostró mayor proliferación de callo, en contraste con el tratamiento 2 que
si bien se obtuvo en un tiempo mínimo la cantidad fue menor. Por tal motivo se tomó la
decisión de realizar una segunda inducción de este tratamiento, para poder utilizarlos en
la transformación de Jatropha.
Figura 3.24. Formación de callos y brotes in vitro de J. curcas con tratamiento de BA+ IBA 2.22 Y
4.92 µM) obtenidos de diferentes explantes. A. Hipocotilo; B. Hoja; C. Hoja cotiledonar; D. Plántula
completa parte radical.

82
Figura 3.25. Formación de callos y brotes in vitro de J. curcas con tratamiento de BA+ IBA+KIN
(4.44, 1.23 Y 2.12 µM) obtenidos de diferentes explantes. A. Hipocotilo; B. Hoja; C. Hoja
cotiledonar; D. Plántula completa parte radical.
De igual manera que se obtuvieron y se regeneraron callos, se obtuvieron embriones
somáticos con el tratamiento de Medio MS + Vitamina del B5 utilizando reguladores de
crecimiento como BA (2.92 µM) y AIA (0.57 µM), obtenidos a partir de hoja cotiledonar
(Fig. 3.26).
Figura 3.26. Embriogénesis somática de J. curcas con tratamiento BA (2.92 µM) y de AIA (0.57 µM)
(MJC2) obtenidos a partir de callo.

83
Además de obtener diferentes estados de desarrollo, con algunas deformaciones
como se observa a continuación (Fig. 3.27).
Figura 3.27. Embriones somáticos en diferentes estadios de desarrollo se observan estadios muy
definidos y otros con algunas deformaciones.
Para ello, también se realizó un análisis del número de embriones generados por
cada matraz, durante el conteo no se separaron los estadios de desarrollo, se realizó
englobando todos los estadios (Fig. 3.28).
Con el tratamiento de MS + Vitaminas del B5 combinando BA (14.762 µM) y AIA
(17.121 µM), utilizando como explante hoja verdadera, se generaron brotes
embriogénicos y en algunos casos organogénesis (Fig. 3.29 y 3.30).

84
Figura 3.28. Respuesta embriogénica de embriones por matraz del tratamiento MJCT2.
Figura 3.29. Regeneración de brotes de J. curcas y obtención de organogénesis.

85
Figura 3.30.Brote embriogénico con 3 meses de crecimiento, se observan algunas esstructura con
forma de cotiledones.
3.5.2. Efecto de los antibióticos sobre el material no transformado
La utilización de construcciones que contienen el gen nptII permite la selección de
las plantas transformadas mediante la adición de un antibiótico aminoglicósido. Para
diseñar los medios con la concentración adecuada de antibiótico es necesario estudiar
previamente su efecto sobre el material vegetal no transformado.
Todos los antibióticos se esterilizaron por filtración y se añadieron al medio
después de la esterilización en la autoclave.
Se realizaron antibiogramas de callos de J. curcas expuestos a diferentes
concentraciones de antibióticos con el fin de conocer a que concentraciones inhibían total
o parcialmente la regeneración en material sin transformar. De esta manera se pretende
establecer la concentración inhibitoria y el antibiótico óptimo para seleccionar plántulas
transgénicas y eliminar o reducir el número de escapes.
Para ello se colocaron los callos en medio de regeneración y se añadieron 0,2.5, 5
y 10 mg/L de Higromicina; 0,25, 50 y 100 mg/L de Kanamicina y 400 mg/L de Cefotaxima
y como testigo el medio sin antibiótico (Fig. 3.31 a 3.33).

86
El resultado fue el siguiente los callos testigo (sin antibiótico) se colocaron en
medio de los demás callos tratados con antibiótico. En la figura 3.31 se observa con
Kanamicina a diferentes dosis en el primer panel se muestra el día cero y el panel
siguiente a los 30 días bajo condiciones de estrés del antibiótico, en el cual se observa
una clara diferencia al testigo al utilizar la dosis letal en este caso 50 mg/L. El medio en el
cual fue colocado fue el medio MS suplementado con vitaminas B5 y con de acuerdo al
antibiótico a evaluar, todo bajo condiciones de fotoperiodo.
Figura 3.31 Efecto de la Kanamicina suministrados a callos de J. curcas en diferentes
concentraciones. Callos en el Día 0 de tratamiento se muestran los callos bien regenerados y sin
daño alguno; Segundo panel. En el centro de cada caja petri con medio MS, se muestra al testigo
(sin antibiotico) y alrededor de el al expueso durante 30 dias, obserbandose un cambio en su
fisiología.
En cuanto a la Higromicina se observa daño celular en la mayoría de los tejidos
expuestos. Sin embargo, se tomó como dosis letal la de 10 mg/L (Fig. 3.32).
Sin embargo, el objetivo de esta parte era obtener un antibiótico que no afectara a
nuestros callos al momento de transformar el tejido con alguna cepa bacteriana, por lo
cual el tratamiento óptimo fue el utilizado con Cefotaxima a una dosis de 400 mg/L, en el

87
cual se observa que no daña el material vegetal y lo conserva de la misma forma que al
testigo (Fig. 3.33).
Figura 3.32. Efecto de la Higromicina suministrados a callos de J. curcas en diferentes
concentraciones. Primer panel. Callos en el Día 0 de tratamiento se muestran los callos sin daño
alguno; Segundo panel. En el centro de cada caja petri, se muestra al testigo y alrededor de el al
tejido expuesto al antibiótico después de 30 días con las concentraciones 2.5 y 10 mg/L la dosis es
letal.

88
Figura 3.33.Efecto de Cefotaxima (400 mg/L) suministrado a callos de J. curcas a los 30 días se
puede observar que los callos no tienen daño celular abundante.
3.6. Desarrollo de protocolo de transformación de callos de J. curcas con
pCAMBIA2301
Una vez obtenido los callos, y contando con la transformación de la bacteria con el
vector pCAMBIA2301, se procedió a realizar el protocolo de transformación de los tejidos.
Pero primero se modificaron algunas variables como: tiempo de incubación, no infiltración
e infiltración al vacío, DMSO (dimetilsulfoxido), dimetilformamida, pH del amortiguador,
solvente para remover la clorofila (etanol y metanol: acetona) y concentración de sustrato.
La prueba histoquímica de GUS fue positiva para los callos que fueron
transformados por agro infección de A. tumefaciens con el plásmido pCAMBIA2301 en
callos tratados sin infiltración al vacio utilizando DMSO a una concentración de 40 mg/ mL
de X-GlcA (Fig. 3.34).

89
Figura 3.34. Localización histoquímica de GUS en callos no embriogénicos de Jatropha sin
infiltración al vacio utilizando una concentración de 40 mg X-Gluc por mL de DMSO. A. Callo en
estadio de desarrollo. B. Acercamiento de tejido teñido. C. callo con malformación a 10x y D.
acercamiento de tejido a 40x.
Se utilizaron diferentes explantes de tejidos para establecer la prueba histoquímica
de GUS, no obstante se encontraron problemas, ya que al momento de infiltrar los callos y
embriones al vacio el tejido se fenolizaba y esto impedía la eliminación de la clorofila.
Para resolver este problema se utilizó en un principio etanol absoluto y etanol al 70% pero
ninguno de los dos podría absorber el componente, por ello se decidió utilizar metanol:
acetona (3:1) esto reportado en la bibliografía y también recomendado por expertos en
este protocolo.

90
Ya teniendo como descartado la utilización del tipo de solvente, pH de la solución
amortiguadora, DMSO y el tipo de explante embriogénico y no embriogénico, se realizó un
diagrama general de los tejidos utilizados como callo embriogénico y no embriogénico,
embriones somáticos y cigóticos, así como el testigo correspondiente a cada uno (Fig.
3.35).
Figura 3.35. Tinción histoquímica de GUS en diferentes tejidos vegetales de J. curcas. A. Callo
verde y blanco, en la primera columna se observa el testigo y en las siguientes los tejidos que
fueron sometidos a la tinción; B. Estructuras embriogénicos en estados de desarrollo; C. Embrión
cigótico utilizando un testigo y los transformados teñidos con el gen reportero uidA.
Así también, se evaluó el efecto de dosis respuesta, utilizando concentraciones de
X-Gluc en los tejidos transformados, obteniendo de esta forma una concentración optima
de 0.02 M (Fig. 3.36).
En resumen, se cuenta con el protocolo general, para callos embriogénicos y no
embriogénicos, los cuales fueron utilizados para el proceso de transformación y
generación del protocolo de tinción de GUS, quedando de manera general: como
disolvente del sustrato de X-Gluc al DMSO a una concentración de 0.02 M, el pH del
amortiguador de 7, el solvente metanol: acetona para desteñir tejidos (Fig. 3.37).

91
Figura 3.36. Dosis respuesta del X-Gluc en cultivos in vitro de Jatropha curcas transformados con
la ceba LBA4404::pCAMBIA2301. A y B. Embriones somáticos utilizando una concentración de
0.02 M (1 mg/mL) de X-Gluc; C y D. Callo verde teñido a una concentración de 0.02 M; E y F.
Embriones somáticos tenidos con X-Gluc a una concentración de 1.8 M (40 mg/mL). El sustrato 5-
bromo-4-cloro-3-indolyl-β-glucorinida fue disuelto con 1 mL de Dimetilsulfoxido utilizando un pH de
amortiguador de sustrato de 7.
Figura 3.37. Protocolo general de transformación de callos embriogénicos de J. curcas. A. Plántula
a 15 días de germinación medio liquido; B. Formación de callo en el tallo de la plántula; C.
Estructuras originadas después de 60 días de inducción (BA+AIA) medio liquido; D. Organogénesis
obtenida a 30 días después de inducción; E. Estructuras embriogénicas con algunas
malformaciones; F. Callo de 2 meses de desarrollo; G. Callo embriogénico transformado con el gen
uidA tratamiento 1 mg/Ml; H-I. Embrión transformado después de 1 mes de teñido.

92

93
CAPÍTULO IV
DISCUSIÓN GENERAL
En la presente tesis se logró identificar la expresión basal del gen KASII en
diferentes días post-antesis de la semilla, en los cuales se pudo observar un mayor nivel
de expresión en el día 30, esto puede deberse al día de maduración que está teniendo el
tejido y la producción de aceites que se está llevando a cabo.
De forma general, el contenido de ARN por tejidos fue mejor y mayor en semillas
maduras y decreció conforme la edad de madurez, así como en hojas y tallo, sin
embargo, en semillas inmaduras y maduras y decreció conforme la edad de madurez, así
como en hojas y tallo, comparado con lo reportado en el 2012 por Wei et al.
Con respecto al aislamiento completo del gen, se ha logrado obtener diferentes
respuestas de acuerdo a las condiciones de amplificación, no obstante, ya se cuenta con
el amplicon completo, comparado con lo publicado en el 2012, donde obtuvieron un
fragmento pequeño para después obtenerlo completo utilizando el 3´RACE.
Se utilizó callo obtenido con los tratamientos expuestos anteriormente de J. curcas
inoculados con la bacteria de Agrobacterium tumefaciens cepa LBA4404, la cual fue
previamente transformada con un vector binario pCAMBIA1301, este vector contiene el
gen reportero uidA (β-glucuronidasa) y el marcador de selección nptII [59].
Hasta ahora solo se ha reportado un trabajo de sobreexpresión de un gen
relacionado con el aumento o disminución de ácidos grasos en J. curcas, la diferencia de
este proyecto está en la trasformación por medio de Agro infiltración, utilizada como una
herramienta biotecnológica para el mejoramiento genético de Jatropha. No obstante, se
decidió dejar esta investigación hasta la obtención del gen completo, debido al tiempo que
se necesitó para obtenerlo, sin embargo en un futuro se seguirá el proyecto, y de esta
forma realizar transformantes incorporando el gen obtenido y analizar la sobreexpresión
en callos y ver si altera la composición de los ácidos grasos.

94
4.1. CONCLUSIONES
Actualmente las sociedades en la mayoría de los países se encuentran inmersas
en la preservación del medio ambiente, alimentos y no sin menos importancia los
biocombustibles [60]. El mejoramiento genético de J. curcas está a la cabeza de estas
posibles soluciones. Aprovechando las técnicas de ingeniería genética, como en la
resistencia a enfermedades y extremos ambientales se ha convertido en una de las
prioridades para hacer de esta planta un modelo exitoso para la producción de
biocombustibles.
Una de las mejoras en este cultivo, es también, manipular la ruta de biosíntesis de
los ácidos grasos que forman los triacilglicéridos. Modificando el perfil de los ácidos
grasos producidos en las semillas, así como la cantidad que almacenan.
De la información generada en la presente tesis, se pude concluir hasta el
momento lo siguiente:
i. Se evaluó la nivel de expresión basal de los frutos de J. curcas.
ii. Se ha logrado obtener por completo el ADNc del gen JcKASII.
iii. Hasta el momento se cuenta con un sistema de regeneración de callos,
embriones y brotes en J. curcas.
iv. Se ha logrado obtener un protocolo eficiente de transformación de callos vía A.
tumefaciens en cultivos in vitro de J. curcas.
4.2. PERSPECTIVAS.
El aporte de la presente tesis ha sido significativo, como perspectivas derivadas del
presente trabajo se mencionan las siguientes:
I. Determinar la expresión basal en los días de floración de la planta del gen KASII.
II. Evaluar la expresión del gen JcKASII bajo diferentes condiciones ambientales
como estrés abiótico y biótico (frio, sequia, etc.).
III. Estandarizar las condiciones para la utilización de PCR cuantitativo con la finalidad
de evaluar la expresión de otros genes.

95
IV. Obtener el protocolo de sobreexpresión de utilizando vectores como pKANNIBAL y
finalmente pCAMBIA2301 para introducirlo a los tejidos (callos).
V. Generar transformantes que se propaguen y realizarles estudios moleculares. Se
espera que la técnica establecida pueda ser aprovechada para la inserción de
genes de interés como el KASII con fines de incrementar el contenido de ácidos
grasos

96
REFERENCIAS BIBLIOGRÁFICAS
[1] Kegl B: Numerical analysis of injection characteristics using biodiesel fuel. Fuel
2006;85:2377-2387.
[2] Kaushik N, Kumar K, Kumar S, Kaushik N, Roy S: Genetic variability and
divergence studies in seed traits and oil content of Jatropha (Jatropha curcas L.)
accessions. Biomass Bioenerg 2007;31:497-502.
[3] Foidl N, Foidl G, Sanchez M, Mittelbach M, Hackel S: Jatropha curcas L. as a
source for the production of biofuel in Nicaragua. Biores Technol 1996;58:77-82.
[4] Wang Z, Calderon MM, Lu Y: Lifecycle assessment of the economic, environmental
and energy performance of Jatropha curcas L. biodiesel in China. Biomass
Bioenerg 2011;35:2893-2902.
[5] Drapcho C, Walker TH: Biodiesel; in Drapcho CM, Nhuan NP, Walker TH (eds):
Biofuels Engineering Process Technology. New York, McGraw Hill, 2008, pp 197-
240.
[6] Wang Z, Calderon MM, Lu Y: Lifecycle assessment of the economic, environmental
and energy performance of Jatropha curcas L. biodiesel in China. Biomass
Bioenerg 2011;35:2893-2902.
[7] Cronquist A: A botanical critique of cladism. Botanical Review 1987;53:1-52.
[8] Toral O, Iglesias JM, Montes de Oca S, Sotolongo JA, García S, and Torsti M:
Jatropha curcas L., una especie arbórea con potencial energético en Cuba. Pastos
y Forrajes 2008;31:191-207.
[9] Dehgan B: Comparative anatomy of the petiole and infrageneric relationships in
Jatropha (Euphorbiaceae). Am J Bot 1-9-1982;69:1283-1295.
[10] Dehgan B, Schutzman B: Contributions toward a monograph of neotropical
Jatropha: phenetic and phylogenetic analyses. Annals of the Missouri Botanic
Garden 1994;81:349-367.

97
[11] Heller J: Physic Nut. Jatropha curcas L. Promoting the conservation and use of
underutilized and neglected crops. 1. Rome, Italy, Institute of Plant Genetics and
Crop Plant Research, Gatersleben, International Plant Genetic Resources Institute,
1996.
[12] Watt SA, Wilke A: Comprehensive analysis of the extracellular proteins from
Xanthomonas campestris pv. campestris B100. Proteomics 2005;5:153-167.
[13] McDougall GJ: Procedure for selection of cell wall-associated glycoproteins.
Phytochemistry 1997;45:633-636.
[14] Kachroo A, Kachroo P: Fatty acid-derived signals in plant defense. Annual Review
Phytopathology 2009;153-176.
[15] Williams M, Francis D, Harwood JL: Lipid biochemistry of tissue cultures of oil-seed
rape. Biochem Soc Trans 1990;18:655-656.
[16] Windsor B, Roux SJ, Lloyd A: Multiherbicide tolerance conferred by AtPgp1 and
apyrase overexpression in Arabidopsis thaliana. Nat Biotechnol 2003;21:428-433.
[17] Wei Q, Li J, Zhang L, Wu P, Chen Y, Li M, Jiang H, Wu G: Cloning and
characterization of a b-ketoacyl-acyl carrier protein synthase II from Jatropha
curcas. J Plant Physiol 15-5-2012;169:816-824.
[18] Sasaki Y, Hakamada K, Matsuno R, Furusawa I, Nagano Y, Suama Y: Chloroplast-
encoded protein as a subunit of acetyl-Coa carboxylase in pea plant. J Biol Chem
1993;268:25118-25123.
[19] Sasaki Y, Nagano Y: Plant acetyl-CoA carboxylase: structure, biosynthesis,
regulation, and gene manipulation for plant breeding. Bioscience Biotechnology,
and Biochemistry 2004;68:1175-1184.
[20] Harwood JL: Fatty acid metabolism. Annu Rev Plant Physiol Plant Mol Biol
1988;39:101-138.

98
[21] Kodama H, Horiguchi G, Nishiuchi T, Nishimura M, Iba K: Fatty acid desaturation
during chilling acclimation is one of the factor involved in conferring lowtemperature
tolerance to young tobacco leaves. Plant Physiol 1995;107:1177-1185.
[22] De Palma M, Grillo S, Massareli I, Costa A, Balogh G, Vigh L, Leone A: Regulation
of desaturase gene expresion changes in membrane lipid composition and freezing
tolerance in potato plants. Mol Breed 2008;21:15-26.
[23] Costa G, Cardoso K, Del Bem L, Lima A, Cunha M, de Campos-Leite L, Vicentini
R, Papes F, Moreira R, Yunes J, Campos F, Da Silva M: Transcriptome analysis of
the oil-rich seed of the bioenergy crop Jatropha curcas L. BMC Genomics
2010;11:462.
[24] Li J, Li M-R, Wu P-Z, Tian C-E, Jiang H-W, Wu G-J: Molecular cloning and
expression analysis of a gene encoding a putative ß-ketoacyl-acyl carrier protein
(ACP) synthase III (KAS III) from Jatropha curcas. Tree Physiol 2008;28:921-927.
[25] Carlson PS, Rice TB: Genetic engineering and crop improvement. HortScience
1977;12:135-137.
[26] Openshaw K: A review of Jatropha curcas: an oil plant of unfulfilled promise.
Biomass Bioenerg 1-7-2000;19:1-15.
[27] Fairless D: Biofuel: The little shrub that could - maybe. Nature 2007;449:652-655.
[28] Wu J, Liu Y, Tang L, Zhang F, Chen F: A study on structural features in early flower
development of Jatropha curcas L. and the classification of its inflorescences. Afr J
Agric Res 2011;6:275-284.
[29] Wei Q, Li J, Zhang L, Wu P, Chen Y, Li M, Jiang H, Wu G: Cloning and
characterization of a b-ketoacyl-acyl carrier protein synthase II from Jatropha
curcas. J Plant Physiol 15-5-2012;169:816-824.
[30] Jain S, Sharma MP: Biodiesel production from Jatropha curcas oil. Renew Sust
Energ Rev 2010;14:3140-3147.

99
[31] Kumar Tiwari A, Kumar A, Raheman H: Biodiesel production from jatropha oil
(Jatropha curcas) with high free fatty acids: An optimized process. Biomass
Bioenerg 2007;31:569-575.
[32] De Block M, Botterman J, Vandewiele M, Dockz J, Thoen C, Gossele V, Movva
NR, Thompson C, Van Montagu M, Leemans J: Engineering herbicide resistance in
plants by expression of a detoxifying enzyme. EMBO J 1987;6:2513-2518.
[33] Jain S, Sharma MP: Biodiesel production from Jatropha curcas oil. Renew Sust
Energ Rev 2010;14:3140-3147.
[34] Jain S, Sharma MP: Biodiesel production from Jatropha curcas oil. Renew Sust
Energ Rev 2010;14:3140-3147.
[35] Jain S, Sharma MP: Biodiesel production from Jatropha curcas oil. Renew Sust
Energ Rev 2010;14:3140-3147.
[36] Kumar Tiwari A, Kumar A, Raheman H: Biodiesel production from jatropha oil
(Jatropha curcas) with high free fatty acids: An optimized process. Biomass
Bioenerg 2007;31:569-575.
[37] Kumar Tiwari A, Kumar A, Raheman H: Biodiesel production from jatropha oil
(Jatropha curcas) with high free fatty acids: An optimized process. Biomass
Bioenerg 2007;31:569-575.
[38] Kumar Tiwari A, Kumar A, Raheman H: Biodiesel production from jatropha oil
(Jatropha curcas) with high free fatty acids: An optimized process. Biomass
Bioenerg 2007;31:569-575.
[39] Banerji R, Chowdhury AR, Misra G, Sudarsanan G, Verma SC, Srivastava GS:
Jatropha seed oils for energy. Biomass 1985;8:277-282.
[40] de Oliveira JS, Leite PM, de Souza LB, Mello VM, Silva EC, Rubim JC, Meneghetti
SMP, Suarez PAZ: Characteristics and composition of Jatropha gossypiifolia and
Jatropha curcas L. oils and application for biodiesel production. Biomass Bioenerg
2009;33:449-453.

100
[41] Deore A, Johnson T: High-frequency plant regeneration from leaf-disc cultures of
Jatropha curcas L.: an important biodiesel plant. Plant Biotechnol Rep 19-4-
2008;2:7-11.
[42] Ogbobe O, Akano V: The physico-chemical properties of the seed and seed oil of
Jatropha gossipifolia. Plant Food Hum Nutr 1-5-1993;43:197-200.
[43] Hawash S, Kamal N, Zaher F, Kenawi O, Diwani GE: Biodiesel fuel from Jatropha
oil via non-catalytic supercritical methanol transesterification. Fuel 2009;88:579-
582.
[44] Klee H, Horsch R, Rogers S: Agrobacterium-mediated plant transformation and its
further applications to plant biology. Annu Rev Plant Physiol 1987;38:467-486.
[45] Murphy DJ: Molecular breeding strategies for the modification of lipid composition.
In Vitro Cell Dev Biol -Plant 2006;42:89-99.
[46] Murphy DJ: Manipulation of lipid metabolism in transgenic plants: Biotechnological
goals and biochemical realities. Biochem Soc Trans 1994;22:926-931.
[47] Singh S, Zhou XR, Liu Q, Stymne S, Green AG: Metabolic engineering of new fatty
acids in plants. Curr Opi Plant Biol 2005;8:197-203.
[48] Von Wettstein D: From analysis of mutants to genetic engineering. Annu Rev Plant
Biol 2007;58:1-19.
[49] Misra P, Toppo D, Mishra M, Saema S, Singh G: Agrobacterium tumefaciens-
mediated transformation protocol of Jatropha curcas L. using leaf and hypocotyl
segments. J Plant Biochem Biot 2012;21:128-131.
[50] Kumar N, Vijay Anand KG, Sudheer Pamidiamarri DVN, Sarkar T, Reddy MP,
Radhakrishnan T, Kaul T, Reddy MK, Sopori SK: Stable genetic transformation of
Jatropha curcas via Agrobacterium tumefaciens-mediated gene transfer using leaf
explants. Industrial Crops and Products 2010;32:41-47.

101
[51] Purkayastha J, Sugla T, Paul A, Solleti S, Mazumdar P, Basu A, Mohommad A,
Ahmed Z, Sahoo L: Efficient in vitro plant regeneration from shoot apices and gene
transfer by particle bombardment in Jatropha curcas. Biol Plant 1-3-2010;54:13-20.
[52] Zaldívar-Cruz JM, Ballina-Gómez H, Guerrero-Rodríguez C, Berzunza-Aviles E,
Godoy-Hernández G: Agrobacterium-mediated transient transformation of annatto
(Bixa orellana L.) hypocotyls with the GUS reporter gene. Plant Cell Tiss Org Cult
2003;73:281-284.
[53] Li M, Li H, Jiang H, Pan X, Wu G: Establishment of an Agrobacterium-mediated
cotyledon disc transformation method for Jatropha curcas. Plant Cell Tiss Org Cult
22-2-2008;92:173-181.
[54] Qu J, Hui-Zhu M, Wen C, Shi-Qiang G, Ya-Nan B, Yan-Wei S, Yun-Feng G, Ye J:
Development of marker-free transgenic Jatropha plants with increased levels of
seed oleic acid. Biotechonology for Biofuels 2012;5:10.
[55] Ye J, Qu J, Bui HTN, Chua N-H: Rapid analysis of Jatropha curcas gene functions
by virus-induced gene silencing. Plant Biotechnol J 2009;7:964-976.
[56] Chi-Manzanero BH: Aislamiento y caracterización del ADNc correspondiente a la
enzima fitoeno sintasa y su relación en la producción de carotenoides en Tagetes
erecta. Mérida, Yucatán, Centro de Investigación Científica de Yucatán, 2001.
[57] Sato S, Hirakawa H, Isobe S, Fukai E, Watanabe A, Kato M, Kawashima K, Minami
C, Muraki A, Nakazaki N, Takahashi C, Nakayama S, Kishida Y, Kohara M,
Yamada M, Tsuruoka H, Sasamoto S, Tabata S, Aizu T, Toyoda A, Shin-i T,
Minakuchi Y, Kohara Y, Fujiyama A, Tsuchimoto S, Kajiyama S, Makigano E,
Ohmido N, Shibagaki N, Cartagena JA, Wada N, Kohinata T, Atefeh A, Yuasa S,
Matsunaga S, Fukui K: Sequence analysis of the genome of an oil-bearing tree,
Jatropha curcas L. DNA Res 2011;18:65-76.
[58] Wei Q, Li J, Zhang L, Wu P, Chen Y, Li M, Jiang H, Wu G: Cloning and
characterization of a b-ketoacyl-acyl carrier protein synthase II from Jatropha
curcas. J Plant Physiol 15-5-2012;169:816-824.

102
[59] Giménez ACA, Menéndez-Yuffá A, García E: Efecto del antibiótico kanamicina
sobre diferentes explantes del híbrido de café Coffea sp. Catimor. Phyton
1996;59:39-46.
[60] Buyx A, Tait J: Ethical framework for biofuels. Science 29-4-2011;332:540-541.